Biological Unitを考慮した分子構造のロード
タンパク質は1本のアミノ酸ポリペプチド鎖が折り畳まって存在しているのがほとんどですが、タンパク質の種類によっては、多量体の形で初めて安定に存在し機能しうるものもあります。Biological Unit(Biological Assemblyとも呼ばれる)とは、実際に生物の中で機能している構造の状態のことを指します。詳しくはPDBjのウェブサイトの「非対称単位と生物学的単位について」も参照してください。
例としてPDB ID: 1ALK(大腸菌由来アルカリフォスファターゼ)とPDB ID: 1EW2(ヒト由来アルカリフォスファターゼ)を比較してみましょう。各構造ファイルを取り扱うRCSB PDBのウェブサイト( https://www.rcsb.org/structure/1ALK と https://www.rcsb.org/structure/1EW2 )のStructure Summaryタブで画面左の方には、タンパク質の構造とともに、Global SymmetryとGlobal Stoichiometryの情報が書かれています。
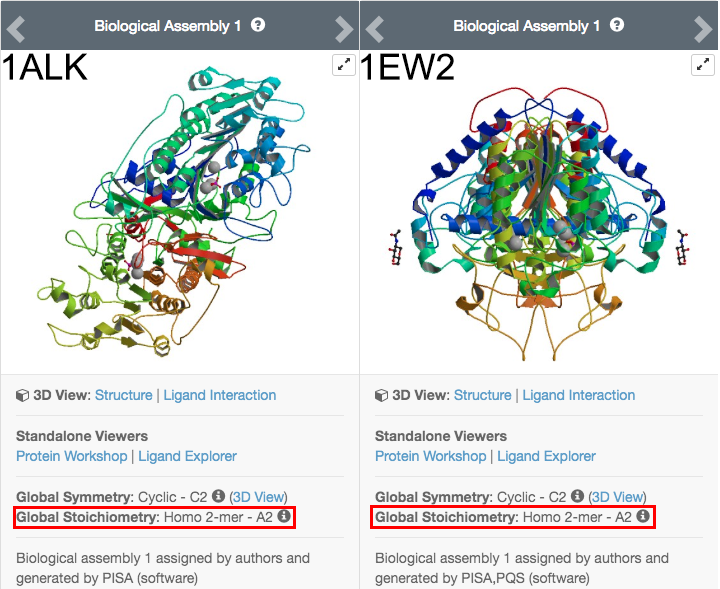
Global StoichiometryにはHomo 2-mer - A2という情報が記されています。論文で調べてみても、このタンパク質がそれぞれホモ2量体として生物中で機能していることが示されています。しかしそれぞれのファイルをPyMOLで単純に開いてみますと、1ALKの方にはA chain、B chainの構造が含まれていますが、1EW2の方ではA chainしか含まれていません。これは、PDBに登録されている原子座標のデータには、必要最小限の「非対称単位」(assymmetric unit)のみ含まれているから です。
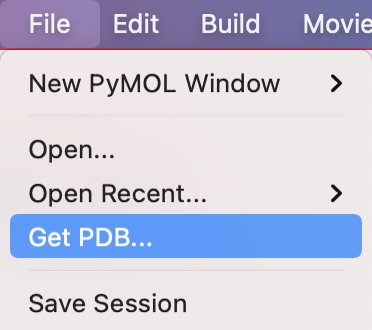
そこで、場合によっては2量体の状態でタンパク質構造を表示してみたいということがあると思います。PyMOL 1.8からはこのBiological Unitを考慮した構造のロードが簡単に行えるようになりましたので、それを1EW2ファイルに対してやってみます。以下の画像のように、[File]->[Get PDB…]を選び、続くウィンドウの画面でIDとAssembly情報を入力します。
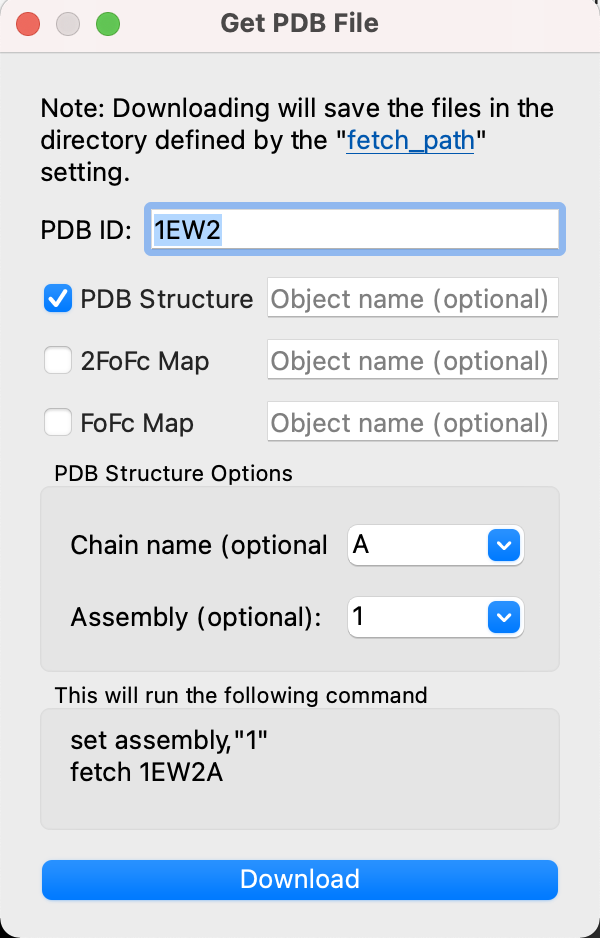
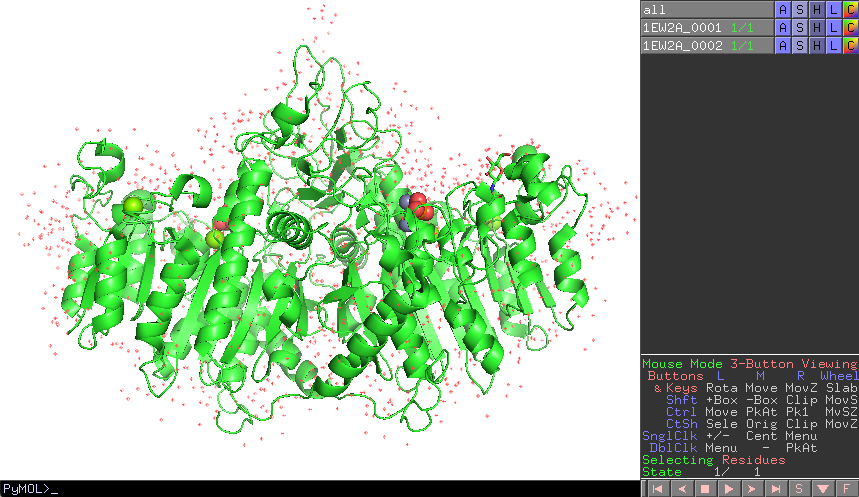
これにより、1EW2の構造がホモ2量体構造で表示されました。
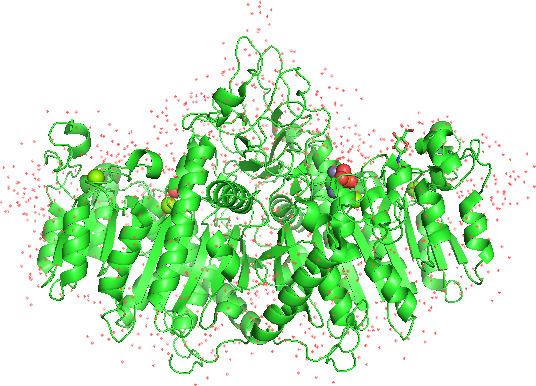
このチェインを別々に扱うためには、右側のオブジェクトパネルから1EW2AのAボタンをクリックし、[state] -> [split]を開いてクリックします。
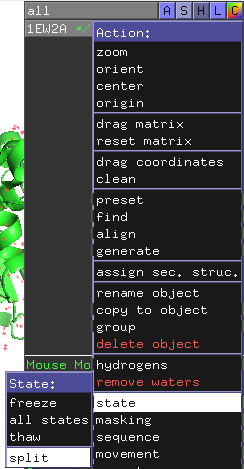
すると、1EW2A_0001, 1EW2A_0002というオブジェクトが新たに生成されます。ここまでくれば1EW2Aオブジェクトは不要ですので、最後に1EW2AのAボタンから[delete object]を押せば完了です。
以上がBiological Unitを考慮した分子構造のロード操作ですが、これは一例であり、他にも同等の操作を達成する方法があります。コマンドラインで上記の操作を達成するには
set assembly, 1
fetch 1ew2
split_states 1ew2
delete 1ew2
でOKです。タイピングが早ければこちらの方が素早くできるでしょう。set assembly, 1に相当する設定変更は、上部メニューの[Setting] -> [mmCIF File Loading] -> [Load Assembly (Biological Unit)]にチェックを入れることでも可能です。