第5章 PyMOLの様々な機能
結晶構造の電子密度マップを表示する
PyMOLは結晶構造の電子密度データを表示させることも可能です。
例としてPDB: 3TG0の大腸菌由来アルカリホスファターゼの電子密度マップを取得して表示させてみます。PDB ID:1ALKのものは電子密度マップがデータベースに登録されていないため、代わりにほぼ同一な構造情報をこちらを例として用います。
# インターネットから3tg0のPDBデータをダウンロード。
fetch 3tg0
# 3tg0の電子密度マップ(2fofc形式)を3tg0_mapというオブジェクトでダウンロードし、表示する
fetch 3tg0, 3tg0_map, type=2fofc
# A chainに結合している無機リン酸PO4に注目する
center /3tg0/H/A/PO4`504
ここまでのロードが終了すると、オブジェクトパネルに3tg0_mapというオブジェクトが追加されているはずです。ここで、続いて上部メニューバーからWizard -> Densityを選択します。
この操作によって、現在の視点周辺に青色の電子密度マップが重なって表示されるようになります。
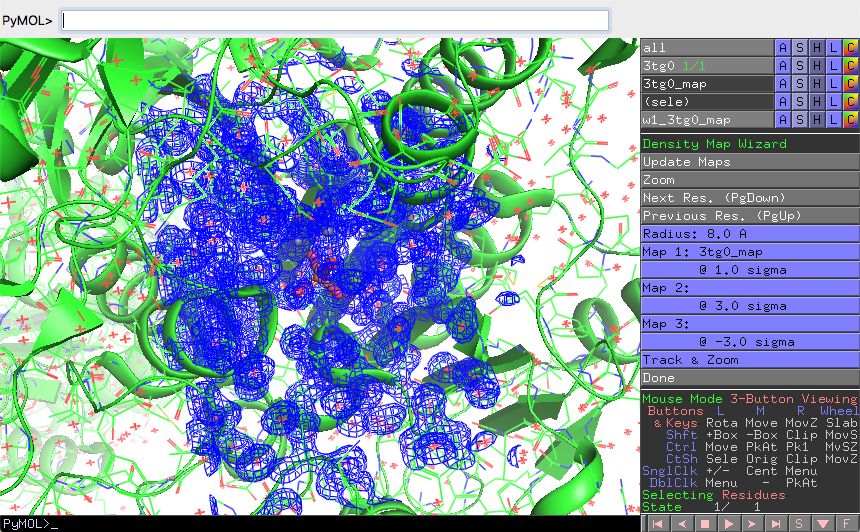
ここで、電子密度マップはw1_3tg0_mapというオブジェクト名になっています。各種表示を変更するには以下の部分をクリックして操作します。
- 色はオブジェクトパネルのCボタンから変更することができます。positiveとnegativeそれぞれについて別の色を指定することができます。デフォルトの青色メッシュは濃くて見えにくいので、もう少し薄い色を指定すると見やすくなるでしょう。
Density Map Wizardと書かれた部分の中のRadiusの部分をクリックすることで、電子密度の表示範囲を変更することができます。@ 1.0 sigmaと書かれているところをクリックすると、電子密度メッシュの表示範囲を変更することができます。sigmaの数字が大きいほど、表示されるメッシュの範囲は小さくなります。- デフォルトではMap 1のみが働いていますが、Map 2、Map 3部分をクリックしてオブジェクト名を指定すれば、さらに重ねて電子密度マップを表示することができます。異なるsigmaのメッシュを色違いで表示させることも可能です。
- 表示設定をやり直したいときは
w1_3tg0_mapのAボタンをクリックし、delete objectをした後、下のWizardのUpdate Mapsを押せば電子密度が再び表示されるようになります。
pair fittingでリガンドの位置を重ねる
ある2つの分子構造があって、一方の分子構造にもう一方の分子構造を重ねたい場合があります。例えば、タンパク質に結合したリガンドの位置を、別の構造に重ねたい場合などです。このような場合に便利なのがpair fittingという機能です。この機能は、PyMOLのWizardメニューの“Pair Fitting“から利用することができます。
1. 2つの構造を用意する
例として、PDB IDが2Z6Fと2O6Pの構造を用意します。2Z6Fと2O6PはそれぞれIsdH-NEAT3 domainとヘムタンパク質の複合体、およびIsdC-NEAT domainとヘムタンパク質の複合体です。2つの構造は似ていますが、IsdH-NEAT3 domainとIsdC-NEAT domainの部分が若干異なります。2つの構造をPyMOL上でダウンロードて読み込みます。ここではそれぞれChain Aだけあればよいので、それだけをfetchコマンドで読み込ませます。
fetch 2Z6FA
fetch 2O6PA
この2つのタンパク質は構造が似ているため、superコマンドを使うことで単純に構造を重ねることができます。
super 2Z6FA, 2O6PA
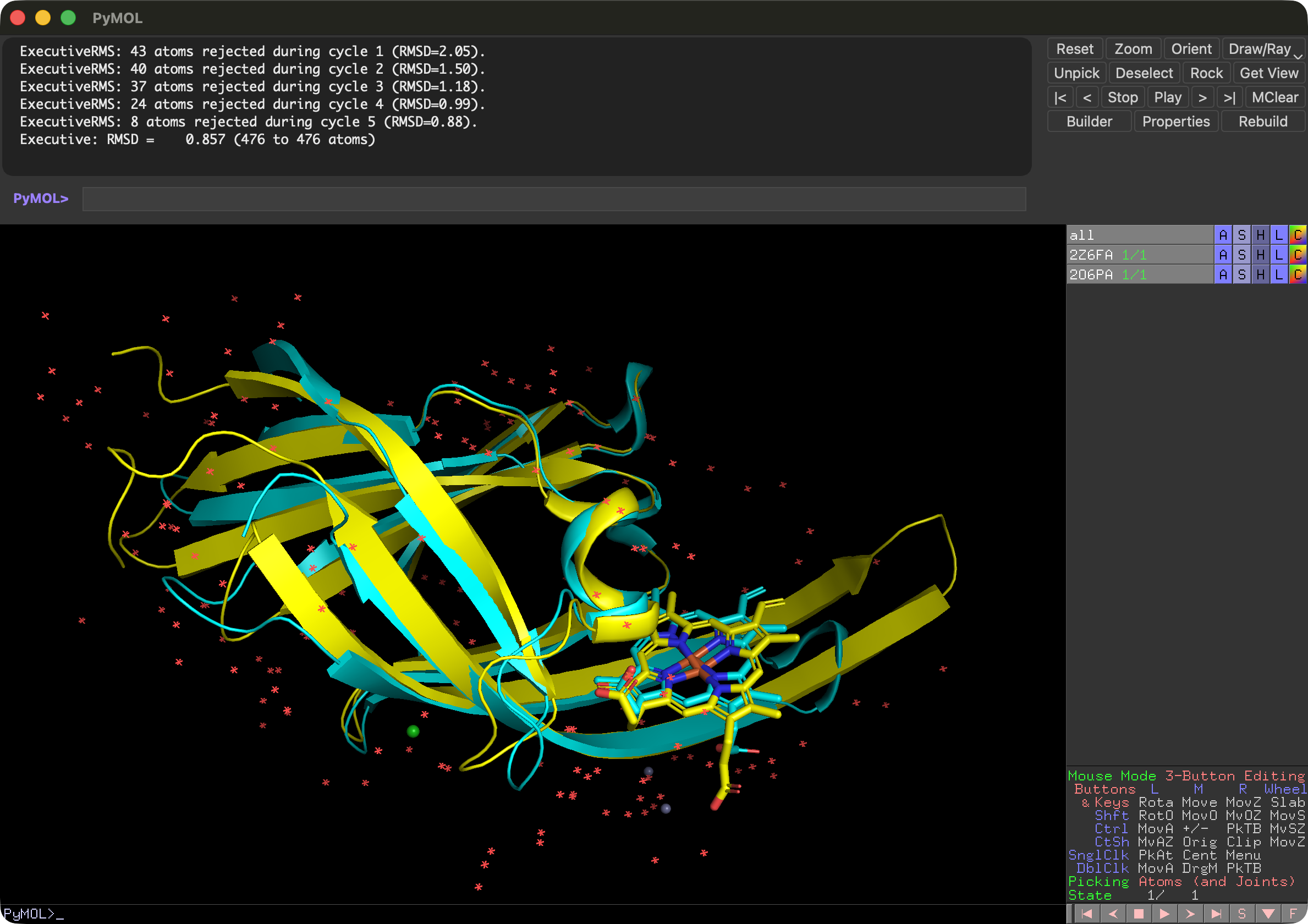
次に、Editing Modeに切り替えて、2つのタンパク質構造を少し離して配置します。

2. pair fittingでリガンドを重ねる
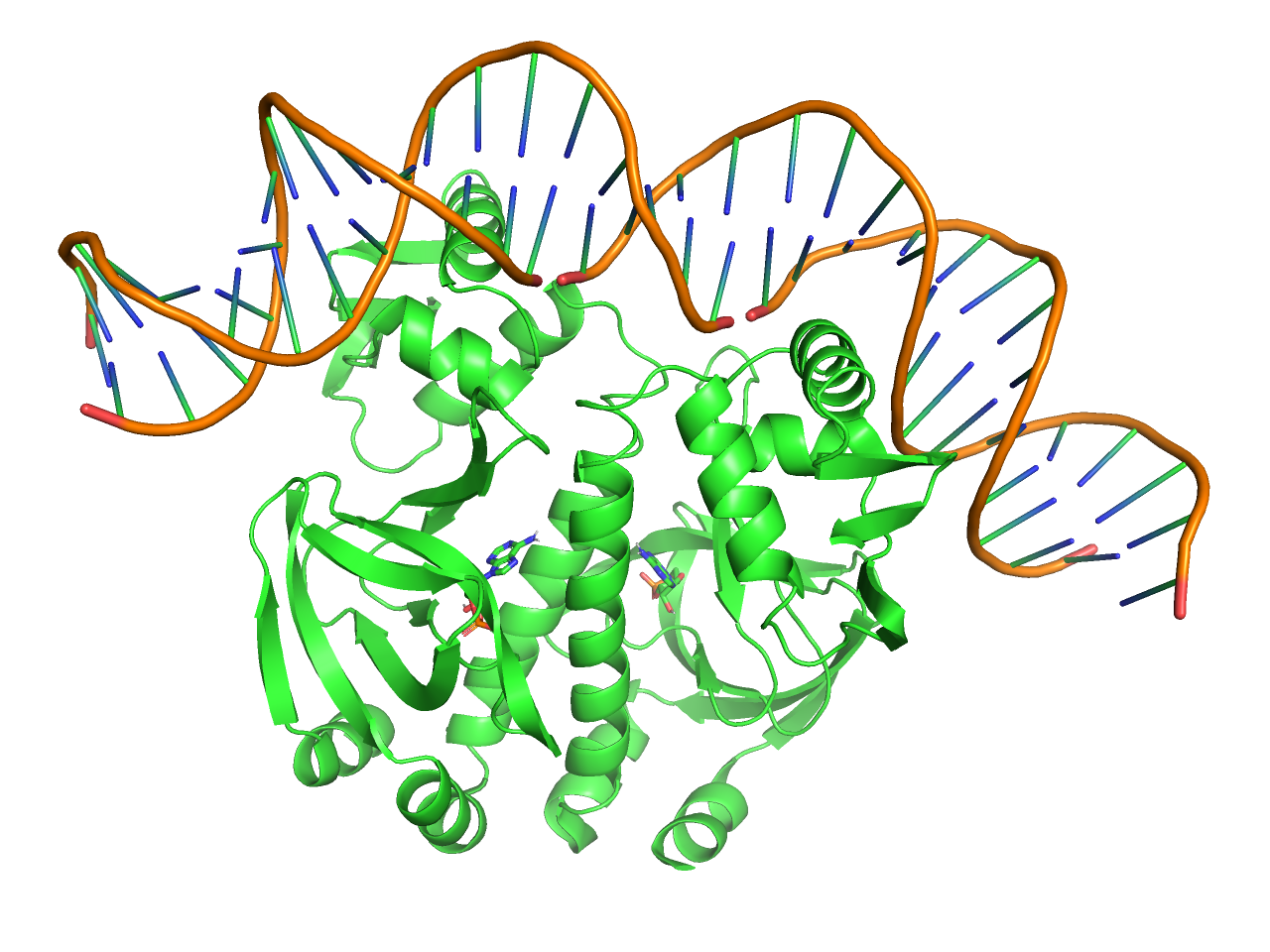
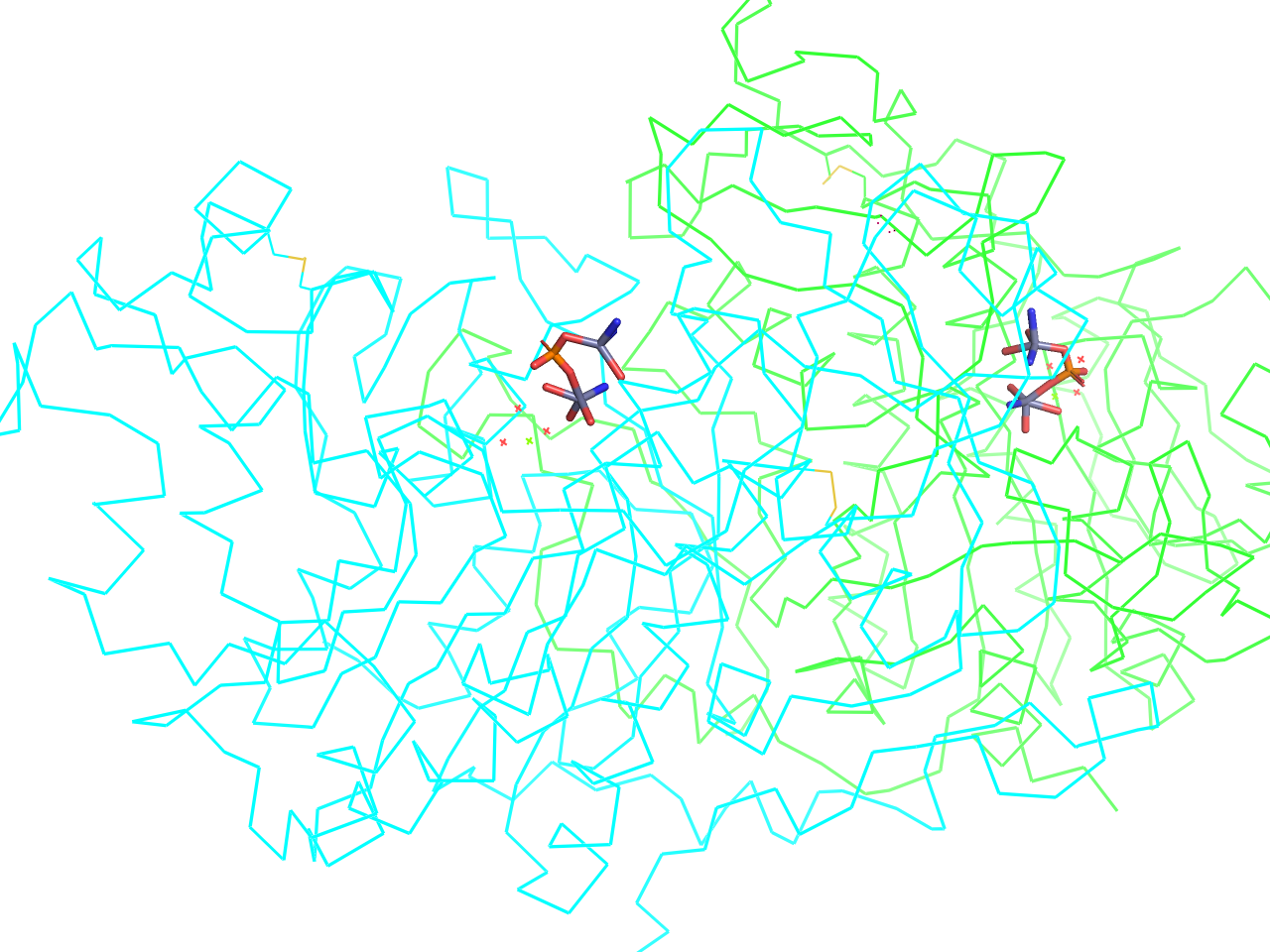
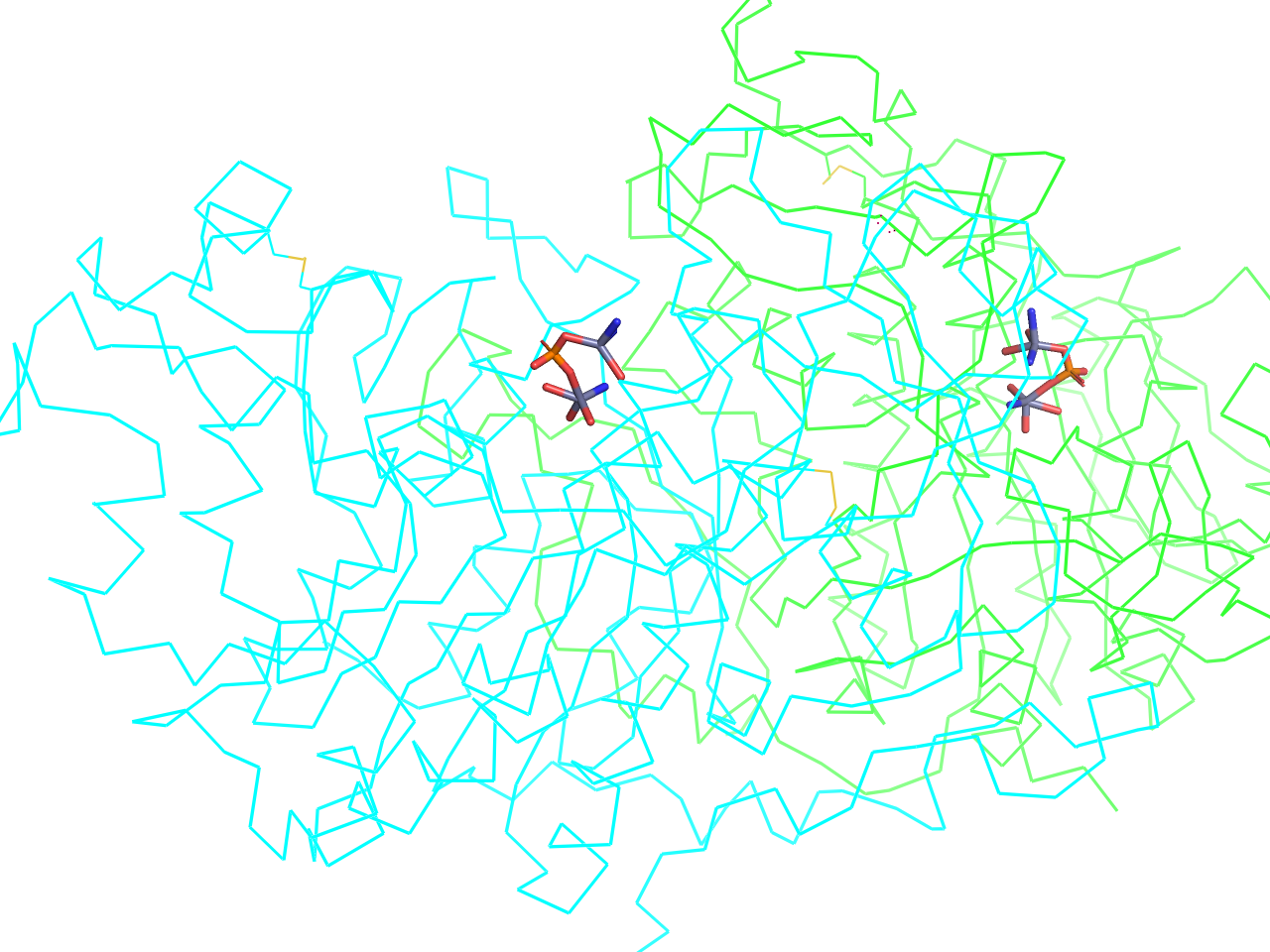
本題として、それぞれの構造に結合しているヘム分子を、タンパク質構造が反転するような形で重ねる操作を行います。
Wizardメニューの Pair Fitting を選択します。このモードでは、2つの分子構造の対応する原子を選択することで、それらの原子を重ねることができます。 モードが始まると、Internal GUIにPair Fittingのメニューが現れ、画面左上には“Pick the mobile atom…“と表示されます。ここで、最初に動かしたい分子にある原子(mobile atom)を選択します。今回は2O6PAのヘム分子を選択します。ヘム分子の原子を1つクリックすると、その原子が選択され、次に“Pick the target atom…“と表示されます。次に、重ね先の分子構造の対応する原子(target atom)を選択します。今回は2Z6FAのヘム分子の対応する原子をクリックします。
今回はヘム分子のポルフィリン環を形成する4つの窒素原子と鉄原子を用いて重ねることにします。4つの窒素原子と鉄原子をそれぞれクリックして選択していきますが、ここで注意が必要なのは、mobile atomとtarget atomの選択を順番に行うことです。つまり、最初にmobile atomの1つ目を選択し、次にtarget atomの1つ目を選択し、次にmobile atomの2つ目を選択し、次にtarget atomの2つ目を選択する、というように交互に選択していきます。これを繰り返して、4つの窒素原子と鉄原子をすべて選択します。選択を間違えた場合は、Internal GUIの Delete Last Pair ボタンを押すことで、最後の選択ペアを削除することができます。
窒素原子を選択する場合は、タンパク質構造を反転させるように位置を選択してください。下の図と動画が参考になると思います。
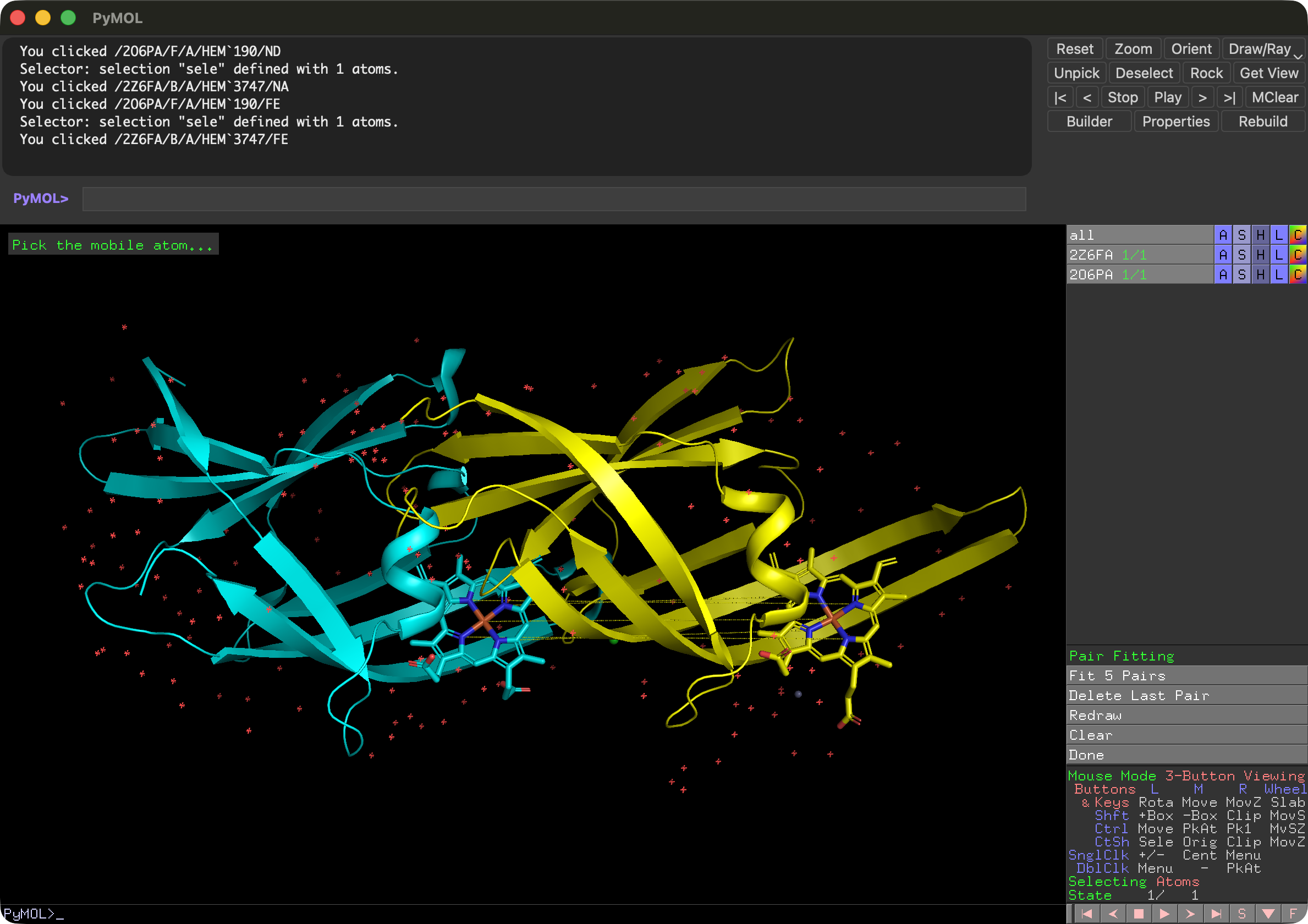
5つの原子ペアを選択し終わったら、Internal GUIの Fit n pairs ボタンを押します(nは選択したペア数)。これで、選択した原子ペアに基づいて、2O6PAのヘム分子が2Z6FAのヘム分子に重ねられます。
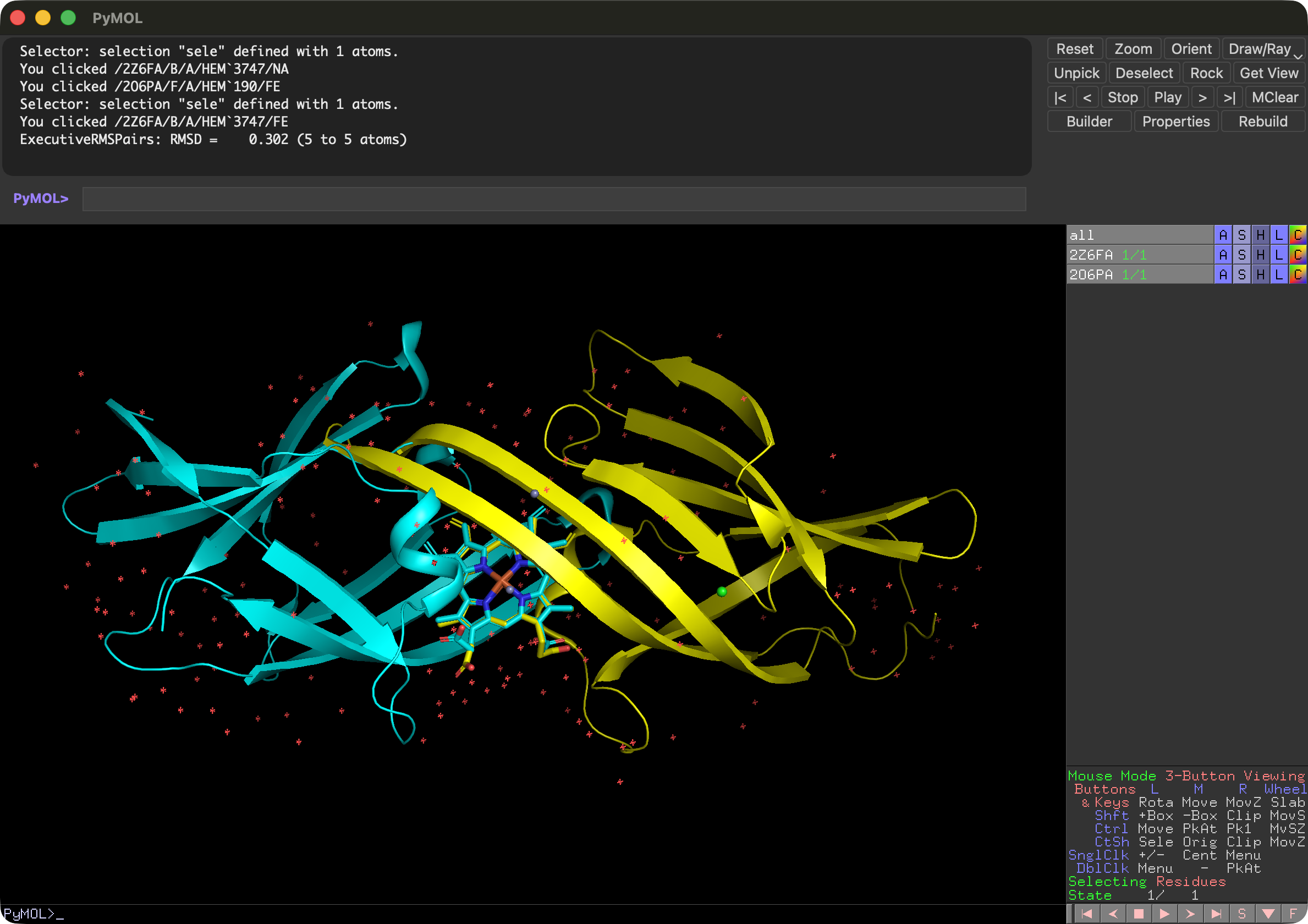
Pair fitting Wizardは、メニューのDoneボタンを押すことで終了できます。
考察
この操作自体はタンパク質構造の重ね合わせと似ていますが、pair fittingでは特定の原子ペアを指定して重ねることができるため、より柔軟に分子構造を配置することができます。特に、Pubchemなどからダウンロードしてきた小分子の分子構造ファイルが、既知のタンパク質-リガンド複合体ポケットに入ることができるかどうかを検討したいときに、そのリガンドの位置に原子をフィッティングさせることで収まりを確認する用途があったり、他にはMDシミュレーションの初期構造としてリガンドの位置を調整したい場合などに有用です。
上記の2Z6F, 2O6Pの例は私が実際に研究で使った例で、IsdH-NEAT3 domainとIsdC-NEAT domainがヘム分子をこのような形で互いに受け渡すことができるのではないか、という仮説を着想するために用いました。実際には、これらを重ねてみるとタンパク質構造どうしがぶつかっているため、そのままではヘム分子の受け渡しができないように思えますが、MDシミュレーションを行ってみると、ヘムとチロシン配位子の軸が少し回転可能なため、ヘム分子の受け渡しが可能であることが示唆されました。
参考:Yoshitaka Moriwaki et al., Rapid Heme Transfer Reactions between NEAr Transporter Domains of Staphylococcus aureus: A Theoretical Study Using QM/MM and MD Simulations, DOI: 10.1371/journal.pone.0145125
APBSプラグインを使った表面電荷表示
PyMOLのプラグインでデフォルトでインストールされているAPBSを使って表面電荷を表示してくれる方法を説明します。
このプラグインは、インストーラー版を使ってPyMOL 2をインストールした場合にはその中に同梱されているので別段の準備は必要ありませんが、オープンソース版をインストールした場合には様々な準備が必要となりますので、下にあるHomebrewでAPBS, PDB2PQRをインストールしておきます。まずはAPBSプラグインの使い方を説明します。
APBSを使ってみる
今回は例としてPDB: 3x2oの構造のみを開いた状態にしておきます。その後、まず上部メニューの[Plugin]を選択し、次に[APBS Electrostatics]を選択します。
中のメニューはこうなっています。
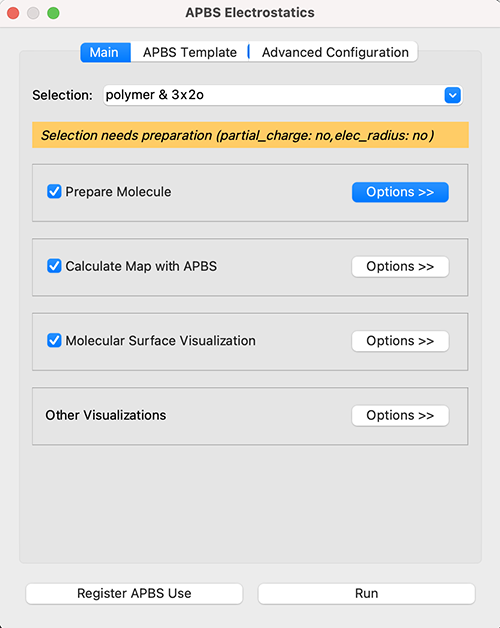
Mainのタブでまずselection部分を確認します。この例ではpolymer & 3x2oという表示になっていますが、これはアミノ酸20種類かつオブジェクト3x2oが選択されていることを意味し、この選択範囲についてAPBSの電荷計算をこれから行うことを意味します。
この選択範囲に問題がなければ、このメニューの下の方にあるRunボタンを押すだけでAPBSの計算が動き出します。途中Warnings: “do you want to continue?“が表示された場合はYesと答えておきましょう。
正常にAPBSの計算が終わると、Close the APBS dialog?と問われます。閉じてもOKです。
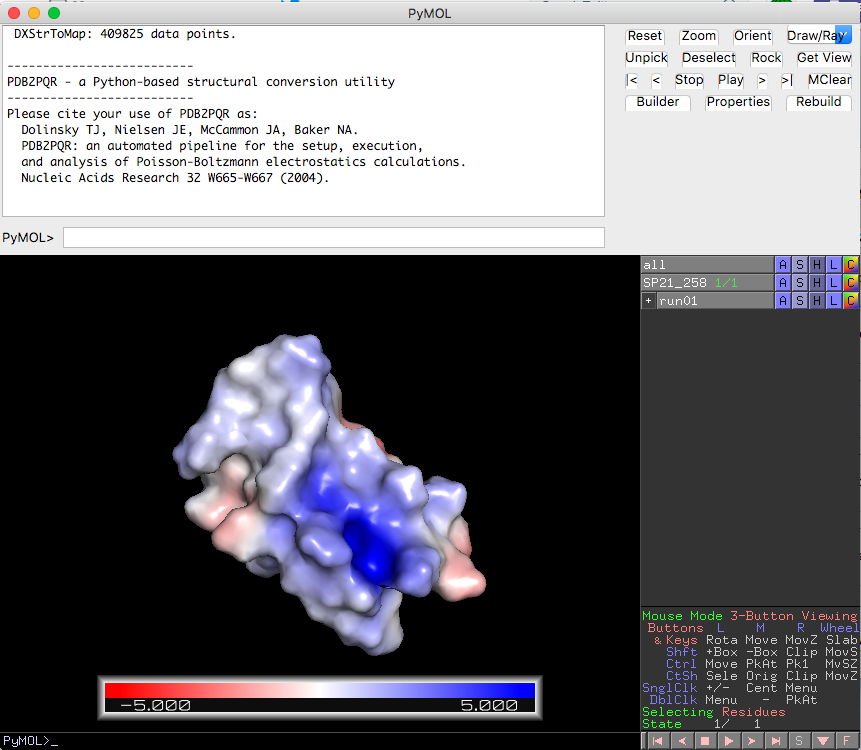
計算が終わると、このように3x2oの表面が赤・白・青に分けて表示されるようになりました。
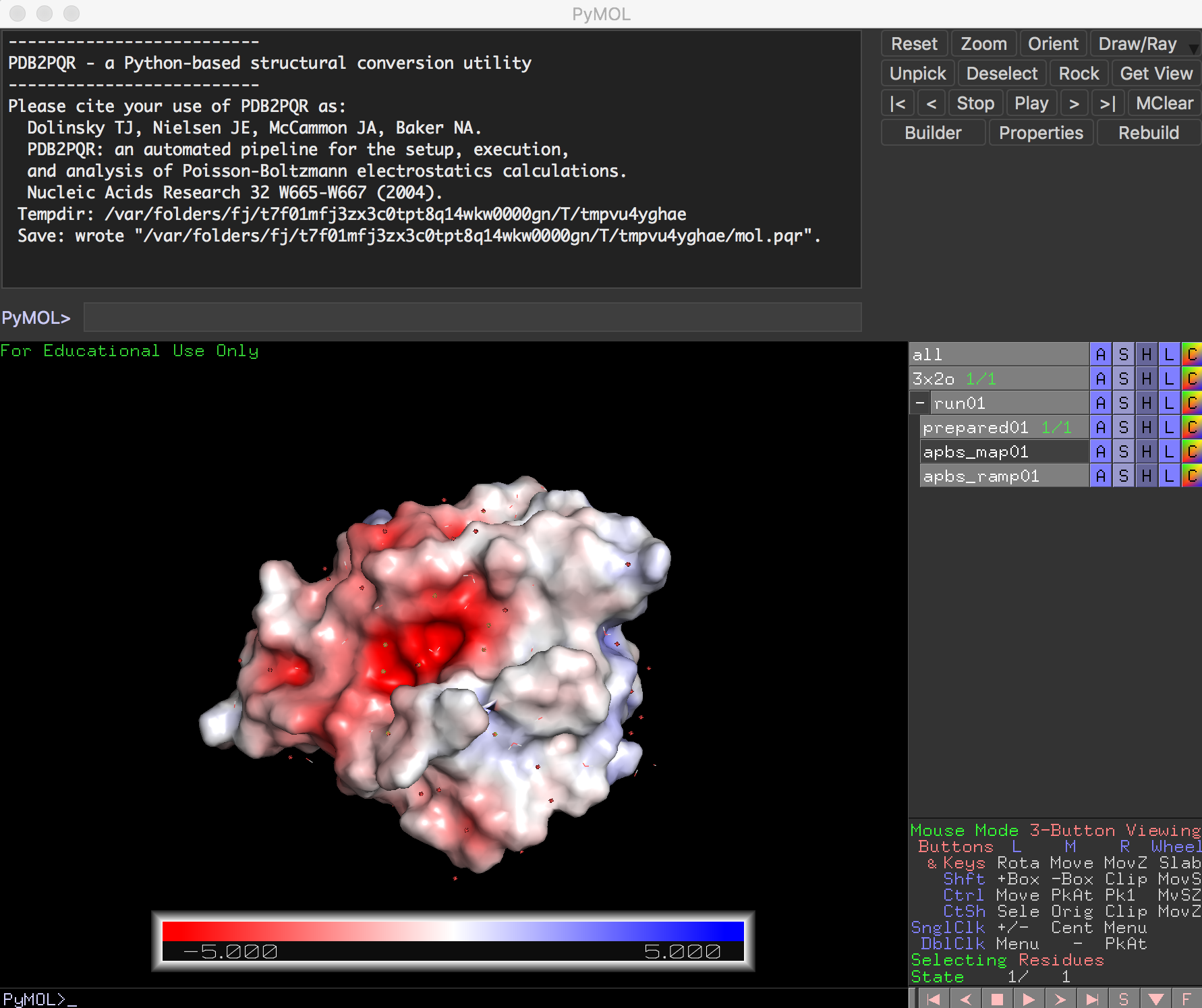
赤は負電荷の多い箇所、青は正電荷の多い箇所を表しています。白は中性です。
右側のInternal GUIのメニューにはrun01というオブジェクトグループが新しく増えています。ここの+マークを押すと子オブジェクトが現れます。このうちapbs_ramp01というオブジェクトのAメニューを開いてみましょう。
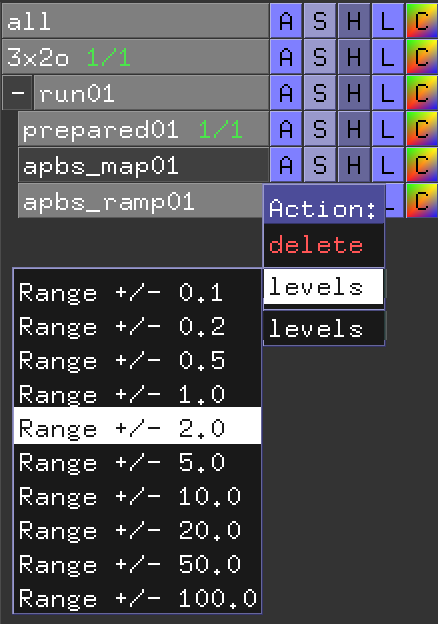
上の方のlevelsを選ぶとRangeを変えることができそうです。このRangeは先述の負電荷・正電荷の色付けの表示領域を表しており、値が小さいほど、描画上で最大となる赤・青のしきい値が小さくなります。つまり、デフォルトの5.0から2.0にしてみると
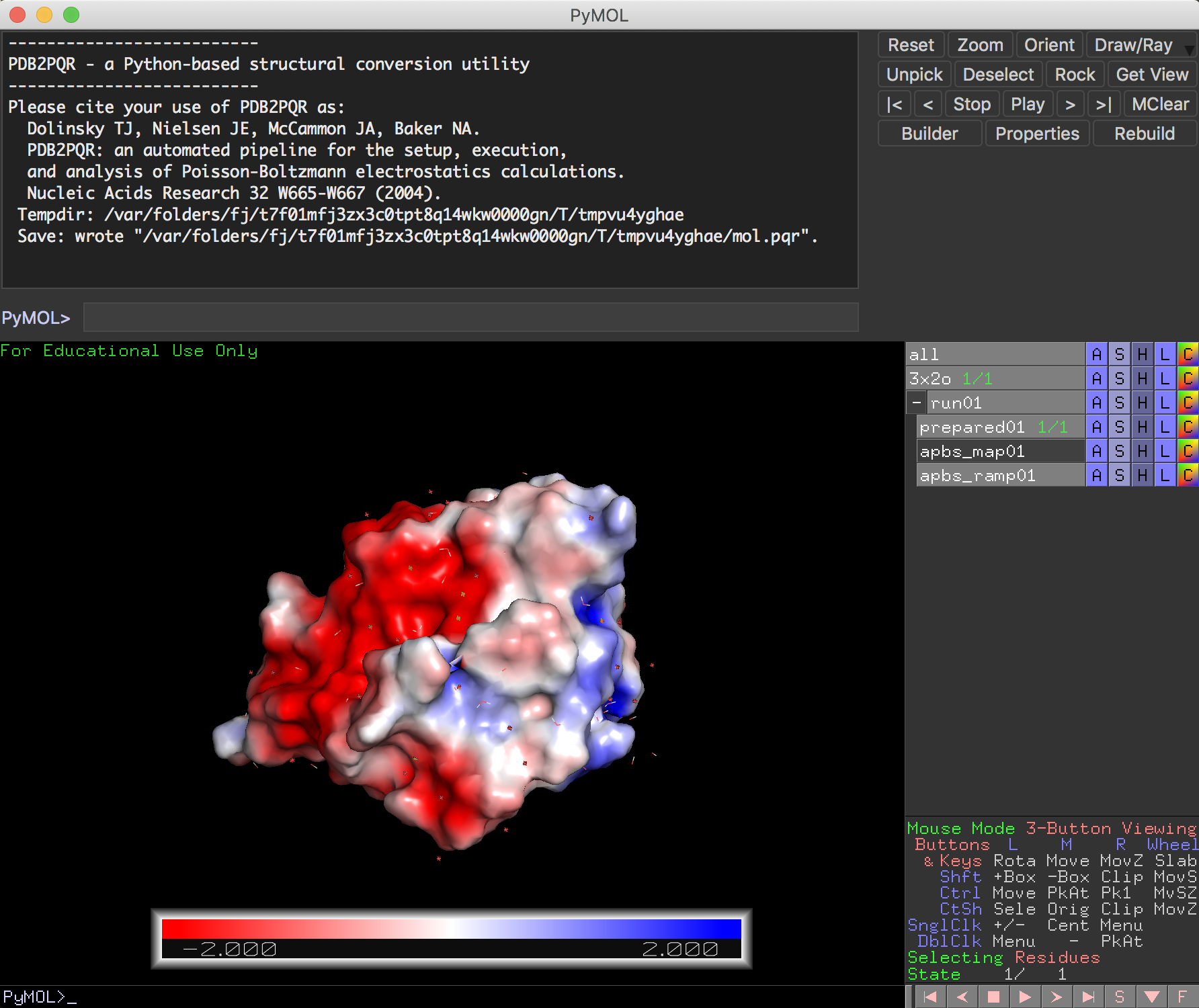
こんな感じで赤と青の箇所が濃く表示されるようになります。
この表面電荷表示はSurface表示として扱われているので、set transparency, 0.5として透明度を変更してあげることができます(0.5は透明度)。
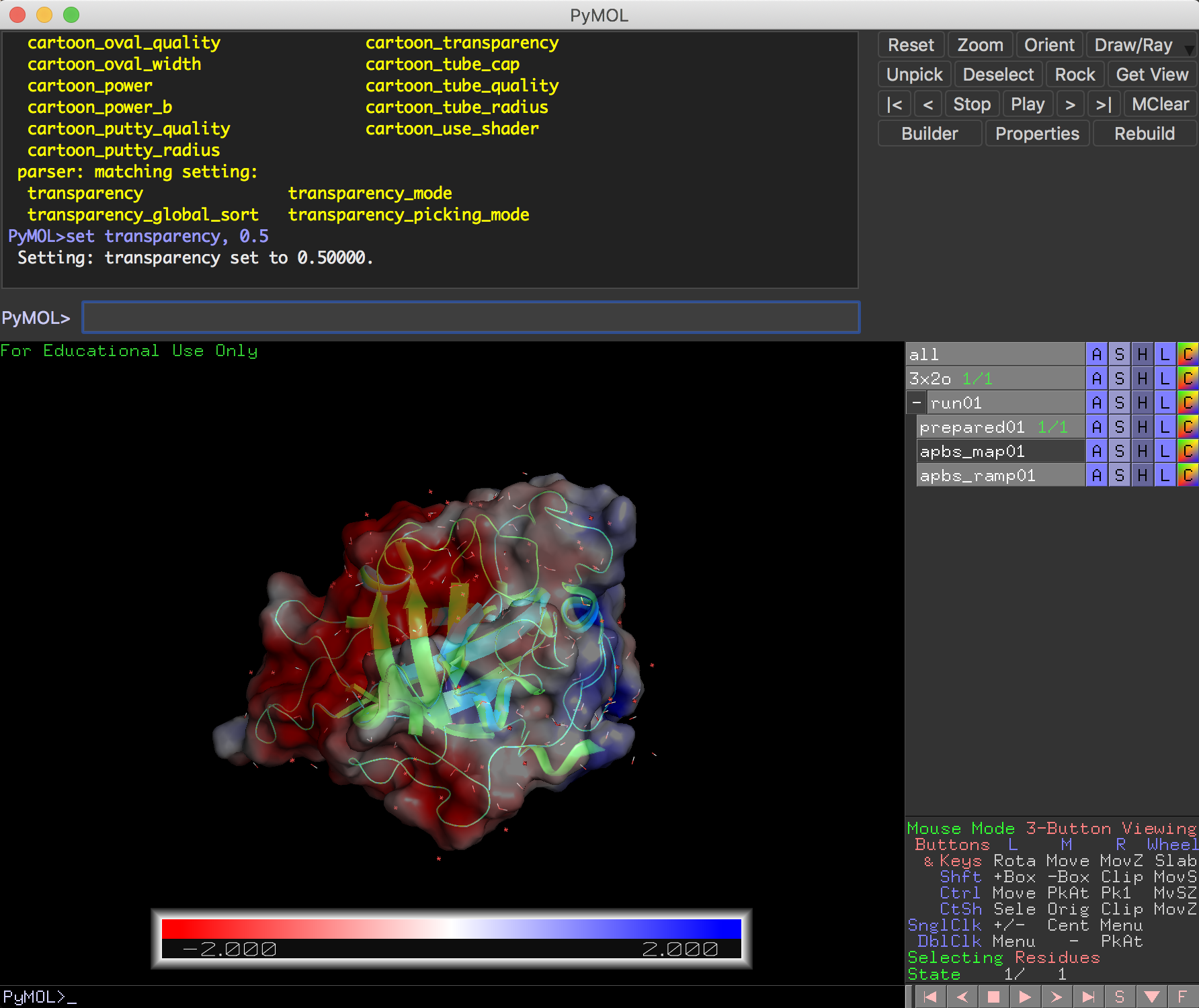
透明度を下げてCartoon表示とともに観察すれば、分子のどの部分構造の付近が負電荷、正電荷になっているかがわかりやすいと思います。
さて、ここで先程のAPBS Electrostaticsメニューのプラグイン画面に戻ってみましょう。そこのMainタブの下の方にはOther VisualizationsというOptionsボタンがあります。ここを開いてみると、「mapオブジェクトが計算されたあと、オブジェクトメニューパネルの“Action“アイテムを使うことで追加の描画を生成することができる」(日本語訳)と書かれてあります。
これに従って追加操作をしてみましょう(書きかけ)。
Advanced Configuration
APBS Electrostaticsの計算のパラメータはデフォルトでも十分問題なく動作すると思いますが、上級者のために、パラメータを変更することもできるようになっています。[APBS Templrate]のタブを開くと、表面電荷を計算するための設定ファイルが表示されます。
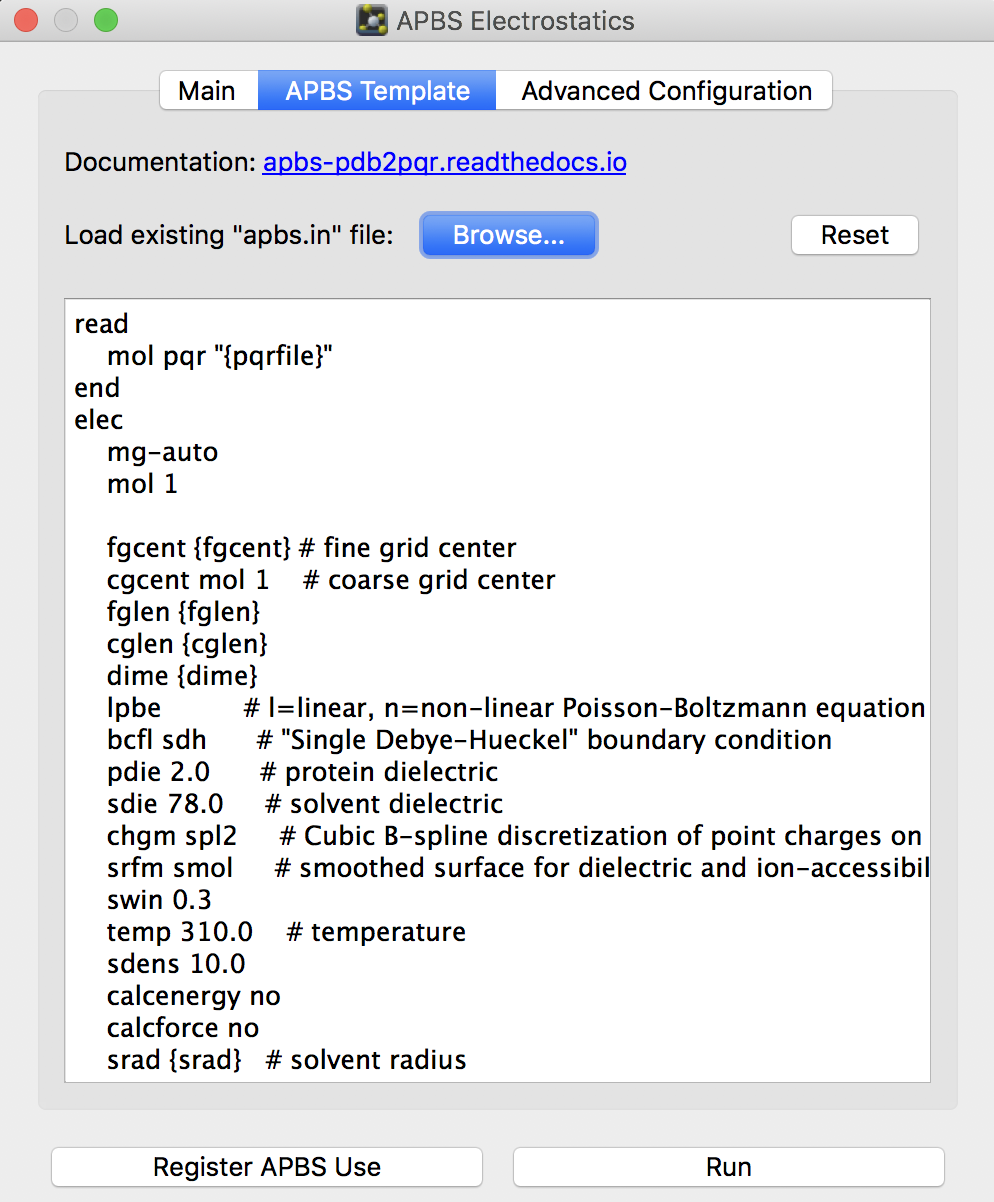
ここのBrowse…のところからは完成された設定ファイルを読み込ませることができます。公式ドキュメントはhttps://apbs-pdb2pqr.readthedocs.io/en/latest/apbs/input/ にあるので、そちらを参考にするのも良いでしょう。
最後の[Advanced Configuration]タブからは、APBSとPDB2PQRのプログラムのインストール場所を指定したり、コマンドオプションを追加して計算させることもできます。追加コマンドオプションの一覧はhttps://apbs-pdb2pqr.readthedocs.io/en/latest/pdb2pqr/invoking.htmlなどが参考になります。
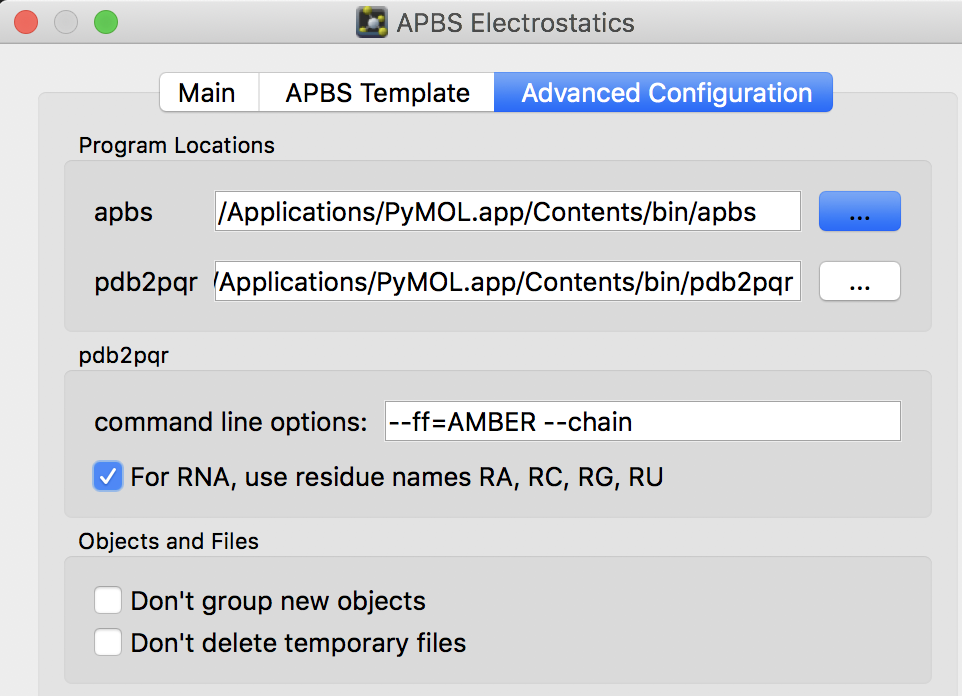
以下の項で説明するように、オープンソース版PyMOLではAPBS, PDB2PQRがプリインストールされていないため、自前で用意してからこのAdvanced Configurationで正しくプログラムの位置を指定してあげる必要があります。
APBSのインストール
(2022年5月3日更新)
オープンソース版PyMOLを使っている人向けの説明です。やり方は3通りあって、Homebrewを使う方法と、バイナリインストールとソースコードからのインストールがあります。macOSの方やLinuxbrewが使える方はHomebrewの方法が簡単です。ソースコードからのインストールの方法は上級者向けです。
macOS(Linux OSの場合はLinuxbrewが必要)を使っている方は、私が作成したHomebrewのFormulaを使うことで簡単にインストールすることができます。執筆時点ではバージョン3.4.0です。
brew install brewsci/bio/apbs
これにより、apbsコマンドが利用できるようになります。
$ apbs --version
----------------------------------------------------------------------
APBS -- Adaptive Poisson-Boltzmann Solver
Version APBS 3.4.1
...
(以下省略)
インストール先はwhich apbsで調べることができます。
$ which apbs
/opt/homebrew/bin/apbs # M1 Macの場合
/usr/local/bin/apbs # Intel Macの場合
ここで表示されたインストール先のパス名をAdvanced ConfigurationのProgram Locationsのapbsの欄に入力すれば動作準備完了です。
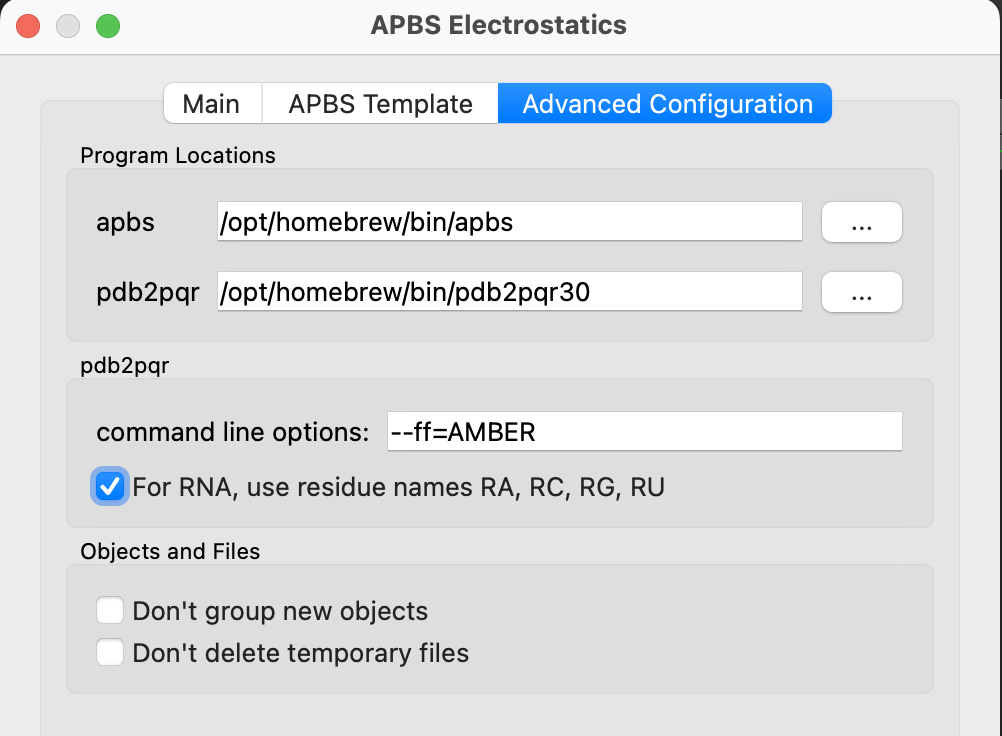
PDB2PQRのインストール
(2022年5月3日更新)
オープンソース版PyMOLを使っている人向けの説明です。pythonのpipを使ったインストールの方法が最も簡単でおすすめです。執筆時点ではバージョン3.5.2です。先に、
$ python3 -m pip install pdb2pqr
...
(中略)
...
Successfully installed pdb2pqr-3.5.2
インストール先はwhich pdb2pqr30で調べることができます。バージョン3以前と異なり、バイナリ名がpdb2pqrからpdb2pqr30に変わっていることに気をつけてください。
$ which pdb2pqr30
/opt/homebrew/bin/pdb2pqr30 # M1 Macの場合
/usr/local/bin/pdb2pqr30 # Intel Macの場合
ここで表示されたインストール先のパス名をAdvanced ConfigurationのProgram Locationsのpdb2pqrの欄に入力すれば動作準備完了です。
APBS GUIプラグインのインストール
オープンソース版PyMOLはバイナリ版と違い、GUIプラグインがあらかじめインストールされていません。しかしバイナリ版のプラグインのディレクトリをそのままオープンソース版の方のディレクトリにコピーしてくれば使用することができます。
このGUIプラグイン部分は私のGithubにコピーして置いてありますので、それをオープンソース版PyMOLのプラグインディレクトリに追加してやります。ターミナルを開いて
# pymolpluginディレクトリをダウンロード
cd ~
git clone https://github.com/YoshitakaMo/pymolplugin.git
# ファイルをオープンソース版PyMOLのプラグインディレクトリに追加
cp -rp ~/pymolplugin/* ${HOMEBREW_PREFIX}/opt/pymol/libexec/lib/python3.14/site-packages/pmg_tk/startup
# ここでPyMOLを立ち上げてみて、プラグインがインストールされていればOK
# インストールできたら~/pymolpluginディレクトリは削除してOK
rm -rf ~/pymolplugin
と入力します。このコピーを行った後にopen-source版PyMOLを立ち上げると、上部メニューのPluginのところにAPBS Electrostaticsの文字が現れているはずです。

この文字をクリックし、Advanced Configurationのタブをクリックします。このProgram Locationsを自身の環境にあわせて設定する必要があります。
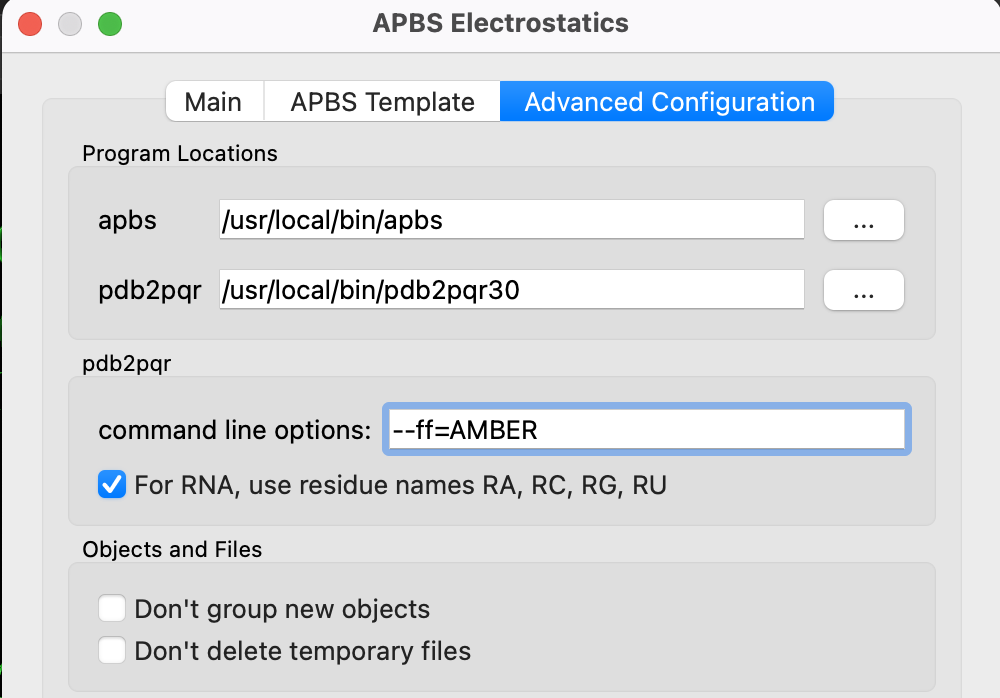
HomebrewでAPBSとPDB2PQRをインストールした場合は、上図のようにapbsの欄を/usr/local/bin/apbsにし、pdb2pqrの欄を/usr/local/bin/pdb2pqr30に設定します(※M1以降のMacの方はそれぞれ/opt/homebrew/bin/apbsと/opt/homebrew/bin/pdb2pqr30)。もしソースコードからインストールした場合にはこの限りではありませんので、適切なapbs, pdb2pqrバイナリ本体へのpathを指定しましょう。
他の部分は特に変更する必要はありません。これで右下のrunを押せばめでたくAPBSが動くはずです。
動画の作成方法
自身の研究しているタンパク質の構造を他の人にもしっかりと見てもらうための方法は、1つにはPyMOLセッションファイルに保存して、それをPyMOLで開いてもらうということが考えられます。しかし、PyMOLセッションファイルは相手のパソコンやmacにPyMOLがインストールされてなければ閲覧することはできません。そこで、より広い範囲で色んな人に見てもらうための方法として、分子が動いている様子を一般的な動画形式に変換するという方法があります。これならPowerpointなどのスライドに埋め込むこともできますし、ウェブ上でも公開できます。
ここではその方法をいくつか紹介します。
目指す動画の例
先に完成例としての動画を表示します。
ポイントは
- 全体像を見せつつ、基質結合部位を見せる
- 背景画像を使う
- 無料のコマンドラインツールのみを使用し、有料アプリを使わない
です。
背景画像を使っているのは、使用しないと黒背景になることが多くなってしまうためです。
使うツール
動画への出力はffmpegを使います。macOSならば
brew install ffmpeg
でインストールできますので、これをインストールしておきます。もしOpen-source版のPyMOLをbrew install pymolでインストールしていた場合はffmpegも自動的にインストールされているはずです。
手順1:画面サイズを決める
動画として出力する予定の画面サイズを先に決めておくと後が楽です。ここでは横1280ピクセル縦720ピクセルの動画を出力することにします。これはコマンドでかんたんに設定することができます。コマンド入力欄に
viewport 1280, 720
と入力すると、画面が指定したサイズに変化します。
手順2:Sceneを保存する
3.7 Sceneで述べているように、Scene機能を使うとPyMOLでのカメラアングルと表示状態を保存することができ、左下に現れたメニューからいつでもその状態を呼び出すことができます。動画を作成する上でもこのScene機能を活用することが可能です。
例としてまずアルカリホスファターゼの二量体構造全体が見える位置でSceneを1つ保存しておきます。いい感じの表示設定とカメラアングルが得られたら、上部メニューのSceneからStoreを選びF1〜F12のいずれかに保存します(ここではF1にしておきます)。
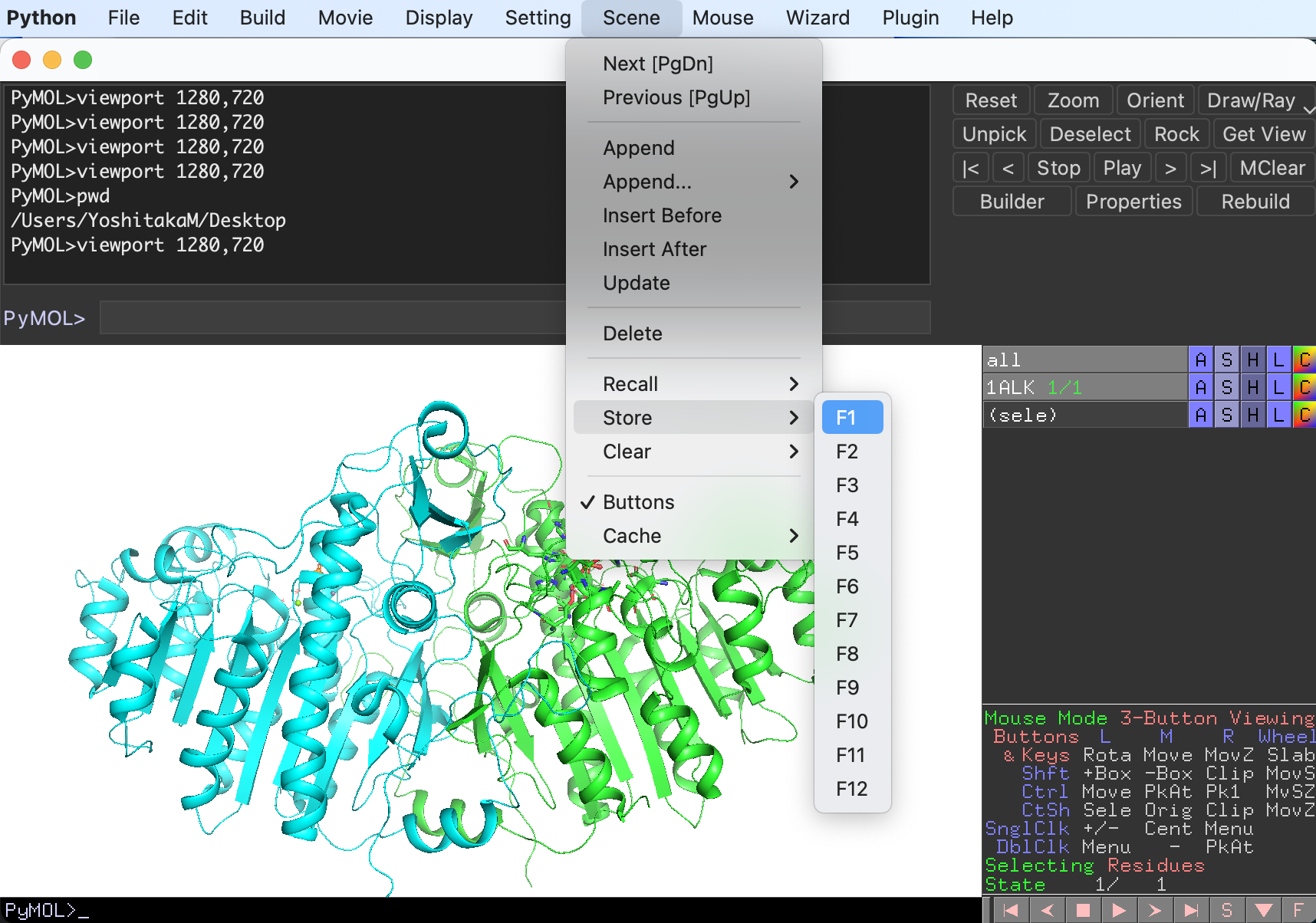
左下にF1ボタンが現れました。以降はこのボタンを押すと表示設定とカメラアングルが呼び出されます。
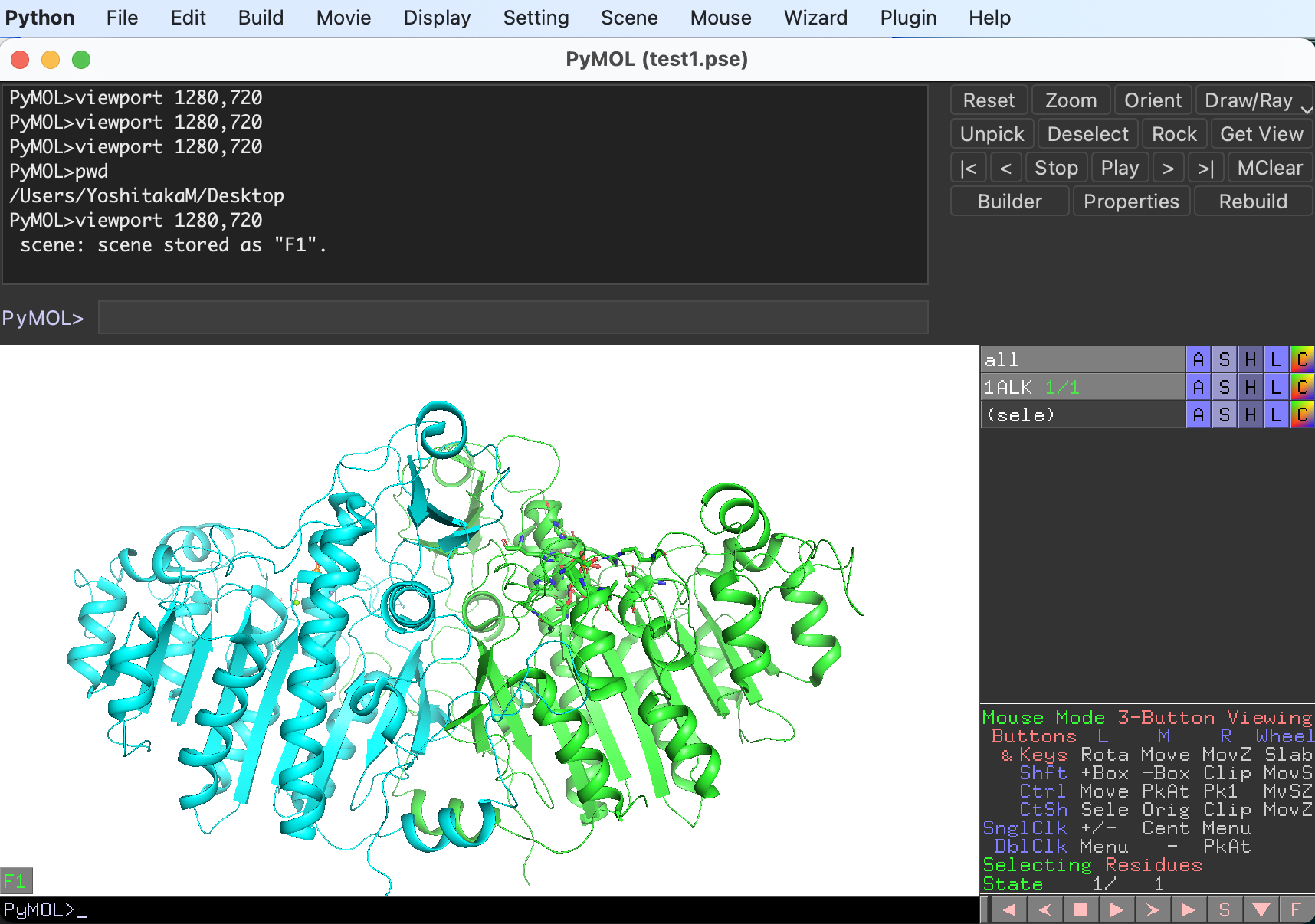
続いて基質結合部位に注目したカメラアングルをSceneのF2として保存しておきます。同様の操作でこんな感じで保存しておきます。
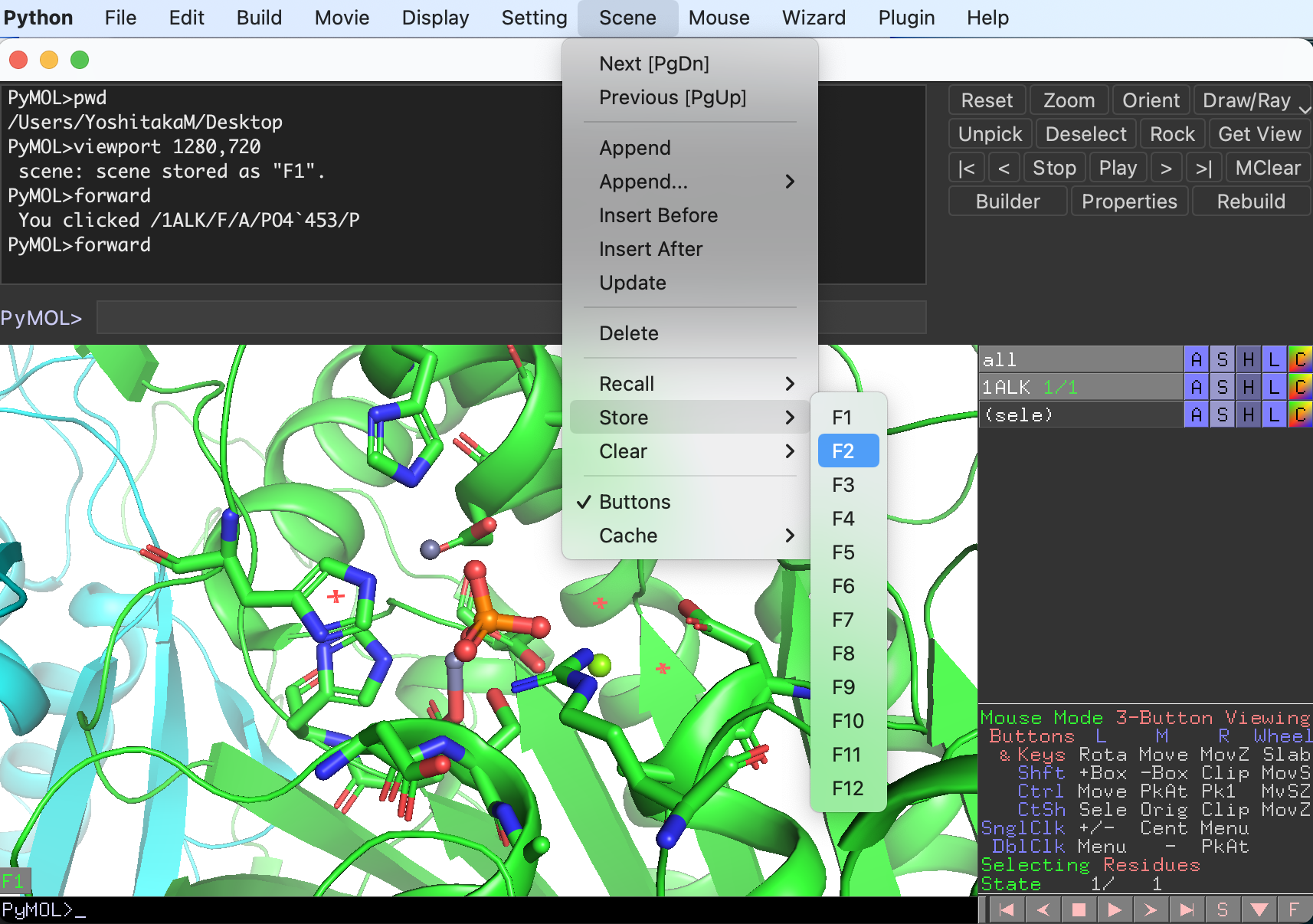
Sceneをすべて設定し終えたら、上部MovieメニューからProgram, Scene Loop, Nutate, 30 de. over 2sec. を選びます。これはSceneを順番に表示しつつ、各Sceneで2秒間30°ずつ揺らしながら次の状態に移行する設定です。設定には色々あるので、3.4 Movieから好きなものを選んでください。
右下のスタートボタンを押すとこんな感じで再生されます。
ここに表示されている画面が動画として出力されるので、よくチェックしておきます。
もし手順1を飛ばしていた場合はここでviewportコマンドを入れて画像サイズを調節し、再度手順2のScene保存をやり直します。
よければセッションファイル(pseファイル)として保存しておきます。
手順3:PNG形式で各フレームを保存する
GUIを使う場合
動画に表示されている動きの全フレームをPNG画像形式で出力させます。これはFile, Export Movie As, PNG Imagesから選択することができます。
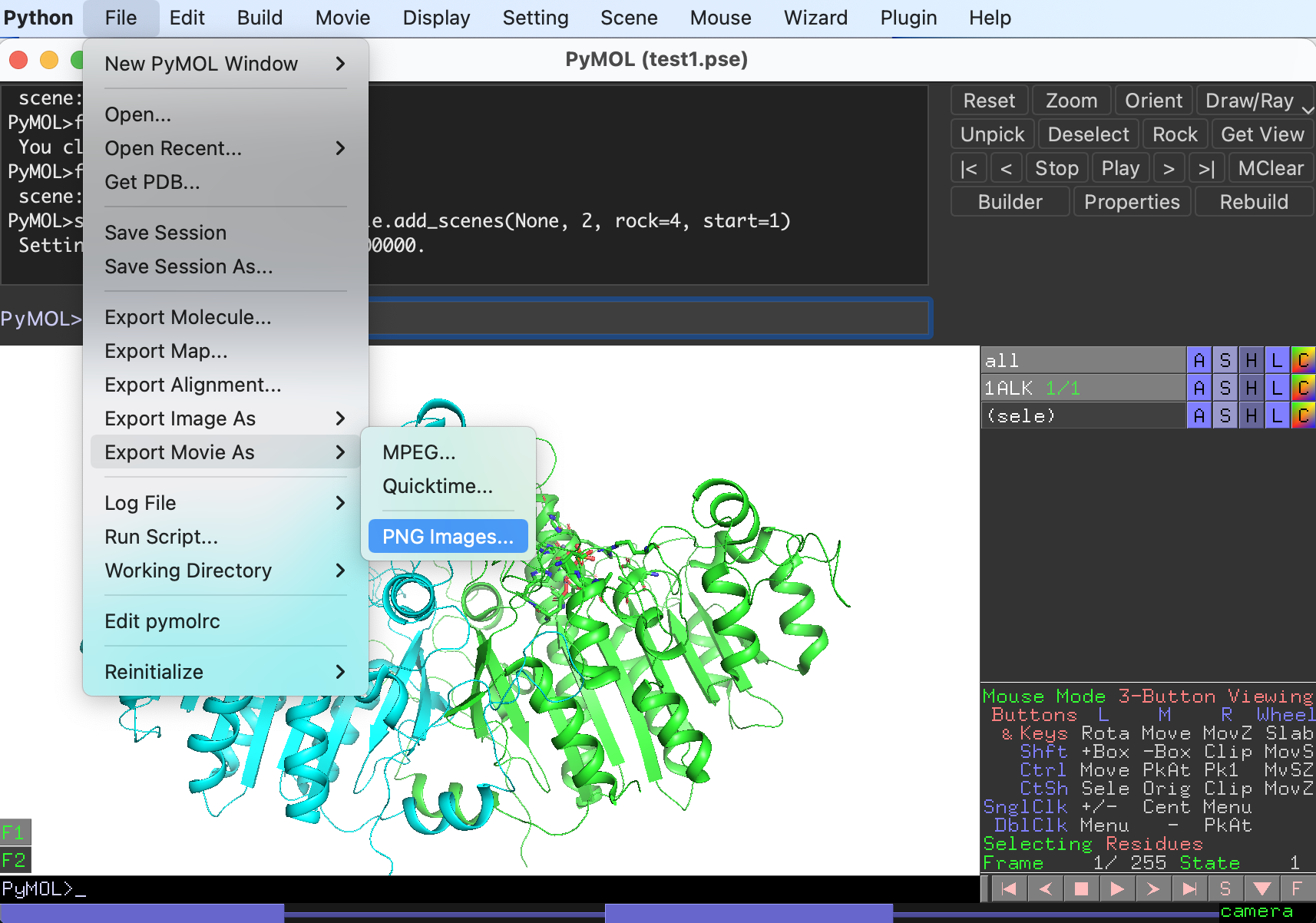
Moview Exportのダイアログボックスでは出力画像サイズを設定することができます。もしここで当初予定していた画像サイズから変わっていれば(少し変わっていることがよくあります)、その数字を修正しておきます。また、RenderingオプションはRay(Slow)の方がきれいに出力されますが、その分時間がかかります。今回はRayでやります。
最後に一番下のSave Movie as…から出力先のディレクトリを選択します。このとき、出力先ディレクトリは新規ディレクトリを作成してからその中に保存することを強く勧めます。こうしないと画像が散らばって面倒です。
今回の例では保存先をmovietestというディレクトリ上に保存することにします。そしてSave Asの名前欄にALPと入力します(この文字は画像のprefixなので任意の文字列でOKです)。
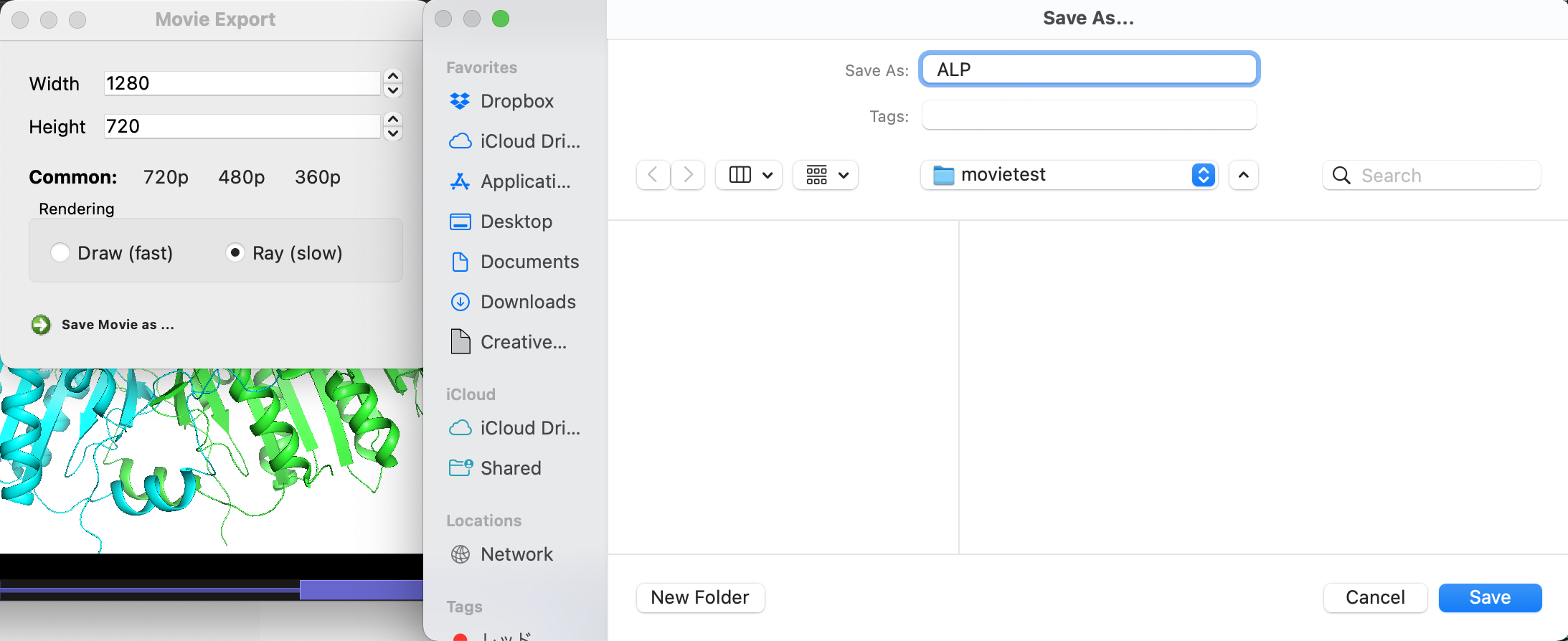
すると、出力先のmovietestディレクトリにALP0001.png, ALP0002.png, … といった具合にすべてのフレームの画像が出力されます。しばらくかかるのでこのまますべて終わるまでじっくり待ちます。
PyMOLのコマンドmpngを使う場合
PyMOLのコマンドラインからmpngコマンドを使うことでも同様にPNG形式で各フレームを保存することができます。例えば以下のようにします。
# syntax
mpng prefix [, first [, last [, preserve [, modal [, mode [, quiet [, width [, height ]]]]]]]]
# example
mpng ALP, first=1, last=120, mode=1, width=1280, height=720
詳しくはPyMolWikiのmpngも参考にしてください。
引数firstとlastを使うことで書き出すフレームを指定できます。これにより、ワークステーションクラスタにレンダリングを自動的に分散させるスマートなPythonプログラムを作成できます。これらのオプションを0に設定した場合、全フレームがレンダリングされます。
prefixにALPを指定すると、出力されるファイル名はALP0001.png, ALP0002.png, … となります。
widthとheightで出力画像のサイズを指定できます。ここで指定することも可能ですが、これを使うと出力される画像を事前に確認することが難しいため、実用上はviewportコマンド(例:viewport 1280, 720)で画面サイズを変更して画面を確認してからmpngを実行する方が良いでしょう。
- 引数
prefix= str: 保存された画像のファイル名のプレフィックス – 出力ファイルには番号が付けられ、.pngで終わります。first= int: 開始フレーム {デフォルト: 0 (最初のフレーム)}last= int: 最後のフレーム {デフォルト: 0 (最後のフレーム)}preserve= 0/1: 存在しないファイルのみを書き込む {デフォルト: 0}modal= int: フレームはモーダル描画ループでレンダリングされますmode= int: 2=ray、1=draw、0=normal {default: -1、ray_trace_framesまたはdraw_framesが設定されている場合はそれに従う}width= int: ピクセル単位の幅 {default: 現在のviewport値}height= int: ピクセル単位の高さ {default: 現在のviewport値}
手順4:ffmpegで動画を生成する
背景画像を使う場合
好きな背景画像を適当に用意します。今回は以下のものを使いました。

画像サイズはあらかじめ出力したタンパク質の画像サイズ(1280 x 720)と合わせておくと良いでしょう。画像の切り抜きにはコマンドラインツールのImagemagickを使うこともできます。詳しくはImageMagick の画像 Cropの記事などを参考にしてください。
この背景画像をbg.jpgとして、先程のmovietestディレクトリに置きます。その後、以下のコマンドでffmpegを実行するだけで、動画ファイルout.mp4が生成されます。
ffmpeg -r 30 -i bg.jpg -vf 'movie=ALP%4d.png [over], [in][over] overlay' -vcodec libx264 -pix_fmt yuv420p out.mp4
-r:フレームレート-i:インプット画像-vcodec libx264:スマートフォン向けの動画(H.264+aac)に変換-pix_fmt yuv420p:エンコーダに渡すピクセルフォーマットを指定。yuv420pはH.264動画変換のときのデフォルトらしい-vf 'movie=ALP%4d.png [over], [in][over] overlay':ALPではじまる連番画像をインプット画像の上に重ねる
これで動画が出力されました。
背景画像を使わない場合
背景画像を使わない場合は以下のコマンドで動画を生成します。
ffmpeg -r 30 -i ALP%4d.png -c:v libx264 -pix_fmt yuv420p out.mp4
連番画像を途中から使いたい場合
連番画像を途中から使いたい場合は-start_numberオプションを使います。例えば11番目の画像から使いたい場合は以下のようにします。
ffmpeg -r 30 -start_number 11 -i ALP%4d.png -c:v libx264 -pix_fmt yuv420p out.mp4
その他
MDシミュレーションでトラジェクトリを表示させたときのタンパク質が動いている様子もこれと同様の手順で出力することができます。
表示形式のプリセット
PyMOLのオブジェクトパネルのAボタンの中にはpresetと言うメニューがあります。これを使うと、様々な表示形式に一発で変換できます。
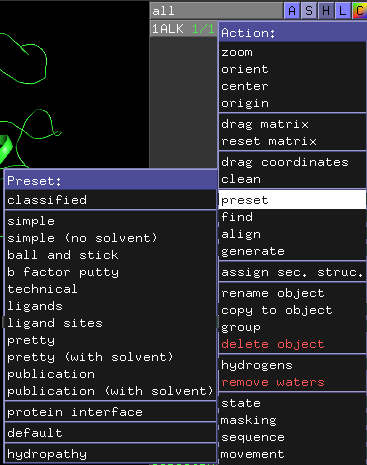
- classified
- simple
- simple (no solvent)
- ball and stick
- b factor putty
- technical
- ligands
- ligand sites
- cartoon
- solid surface
- solid (better)
- transparent surface
- transparent (better)
- dot surface
- mesh surface
- pretty
- pretty (with solvent)
- publication
- publication (with solvent)
- protein interface
- default
- hydropathy
以上のプリセットが用意されています(ver. 2.5.0 時点)。
このプリセットが選択された時、内部処理的には一度デフォルト形式であるclassifiedを設定してから選択されたプリセットの描画設定を上書きしていく形で適用しています。ただしpymolrcに書かれたいくつかの設定値(transparency, surface_quality, surface_type, sphere_scale, stick_radius, stick_color, cartoon_highlight_color, cartoon_fancy_helices, cartoon_smooth_loops, cartoon_flat_sheets, cartoon_side_chain_helperなど)はauto_show_classifiedとしてPyMOL起動時に記憶され、classifiedは描画形式以外この値を利用します。
プリセットの定義は、modules/pymol/preset.py の中に書かれてあります。 https://github.com/schrodinger/pymol-open-source/blob/master/modules/pymol/preset.py も参考にすると良いでしょう。
ここではギャラリー風に紹介していきます。
classified
現在PyMOLのデフォルト設定に最も近い描画形式です。タンパク質構造はCartoon表示、リガンドはSphere表示です。デフォルト設定との細かな違いとして、水分子がwire-nonbonded表示されないなどが挙げられますが、デフォルトの描画設定に戻したいときはこの設定を呼び出すと良いでしょう。
ただし、色設定は変化しないため、手動で戻す必要があります。
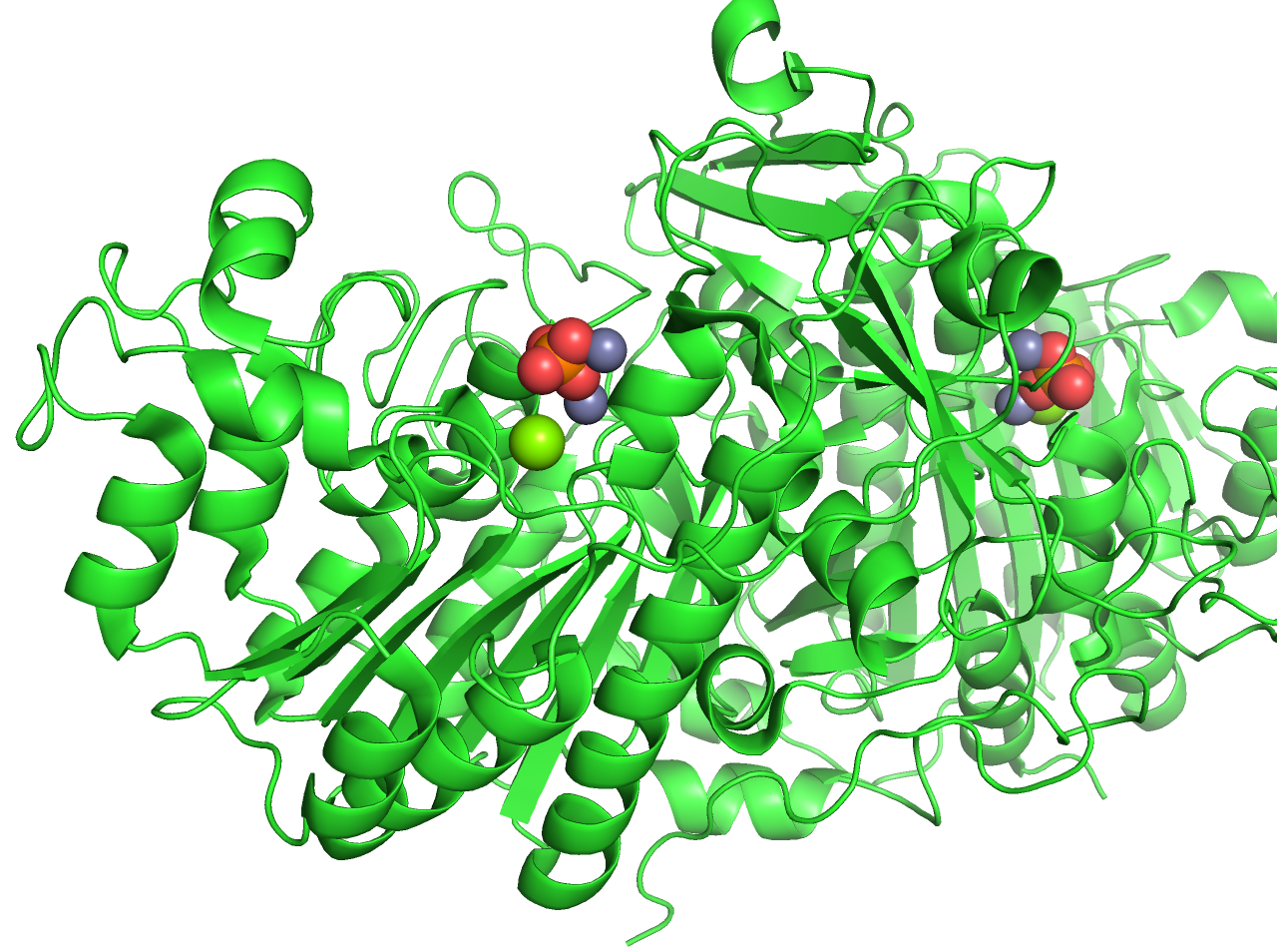
コマンドで行いたい場合は、以下のobjectname部分をオブジェクト名に変えて実行します(例: 1ALK)。以下同様。
preset.classified(“objectname”,_self=cmd)
simple
タンパク質構造は主鎖だけをシンプルに表示するribbon表示、リガンドはStick表示になります。また、チェインごとに自動で色分けがなされます。
preset.simple(“objectname”,_self=cmd)
simple (no solvent)
上記simple表示について、溶媒の表示がなくなったものです。
preset.simple_no_solv(“objectname”,_self=cmd)
ball and stick
各原子を小さめのボールで表し、結合を白色のスティックで表示します。色分けは変化しません。


preset.ball_and_stick(“objectname”,_self=cmd)
b factor putty
温度因子であるB factorをもとに色付けし、さらに温度因子が大きいほど太くチューブ状に表示します。温度因子は青色ほど低く、赤色ほど高くなっています。この表示では主鎖構造のみが表示されます。
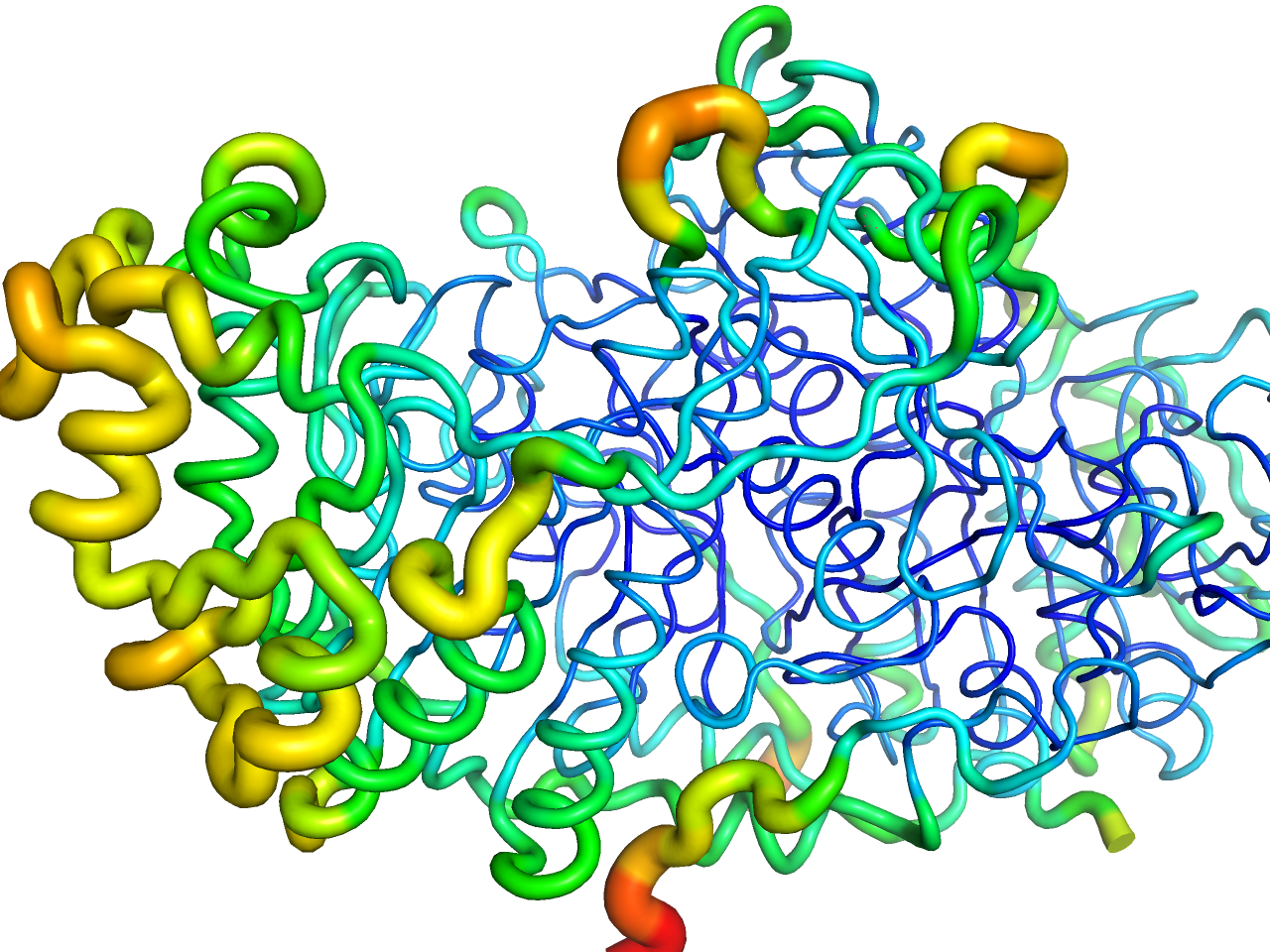
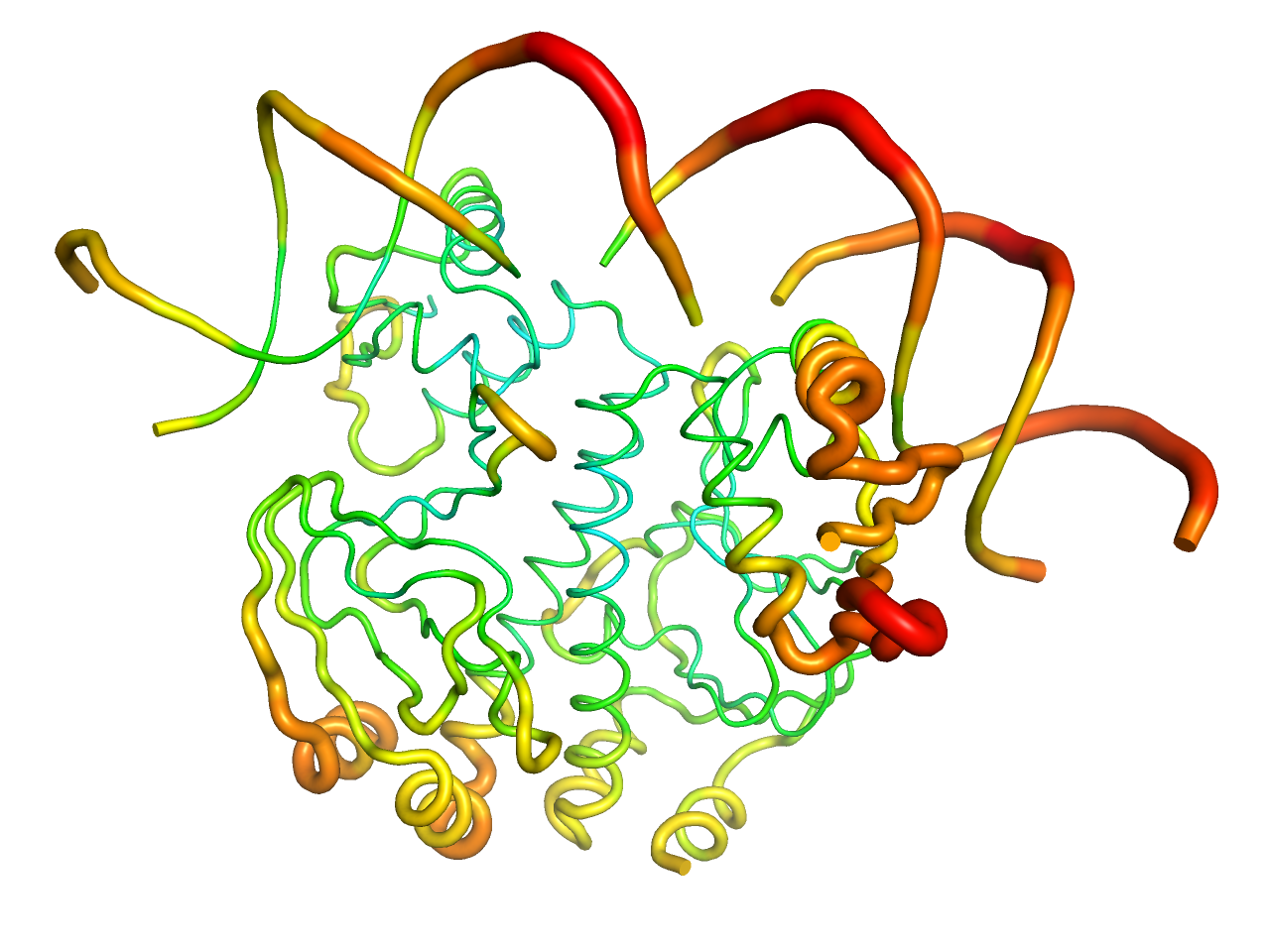
preset.b_factor_putty(“objectname”,_self=cmd)
technical
各チェインのN末端からC末端にかけて青色→赤色となるようなRainbowカラーリングが適用されます。また、水素結合が自動的に検出され、<objectname>_pol_contsというオブジェクトが生成されます。水素結合を表示させたくない場合はオブジェクトパネル上でこの<objectname>_pol_contsの表示をOFFにすればOKです。
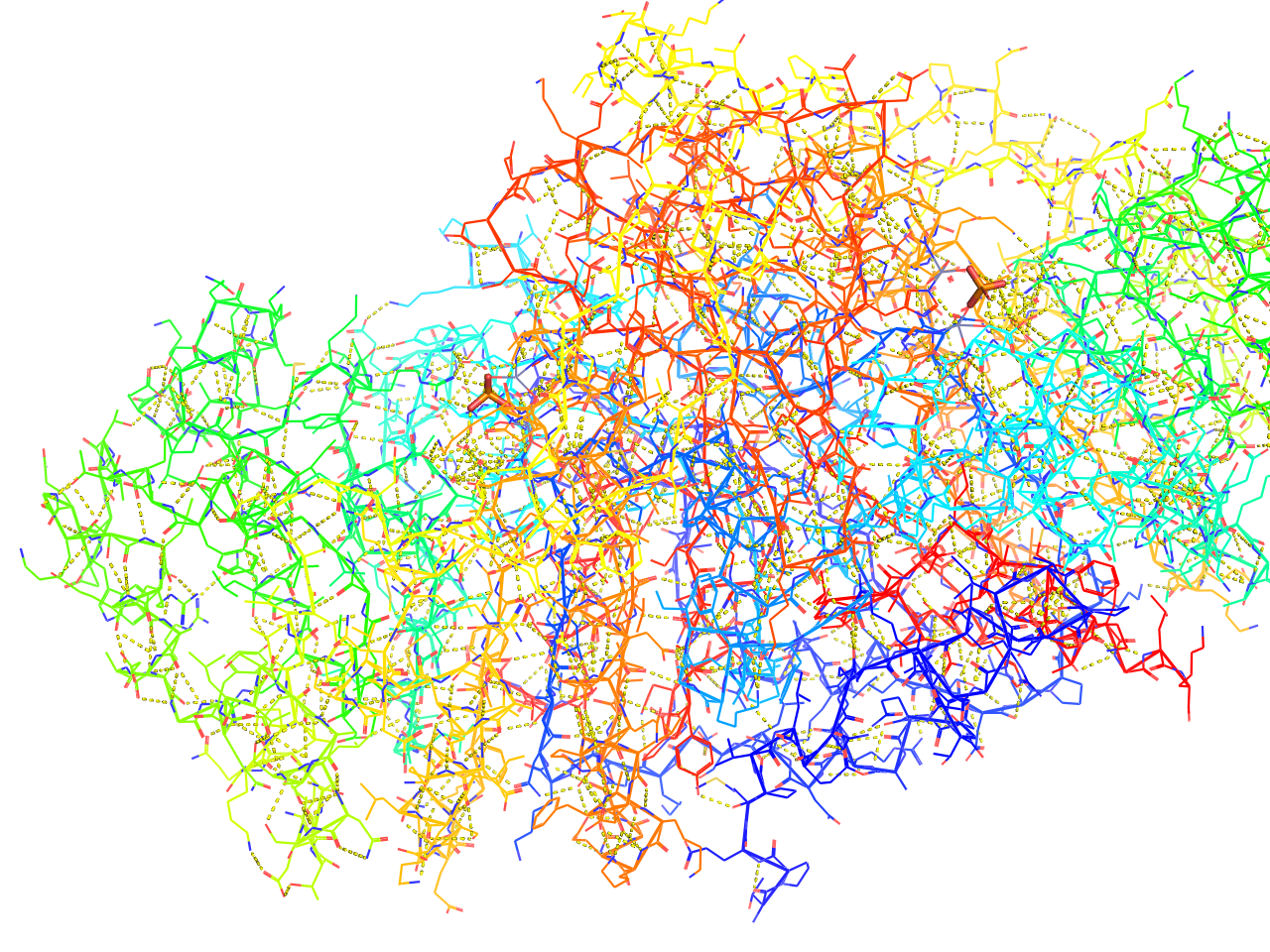
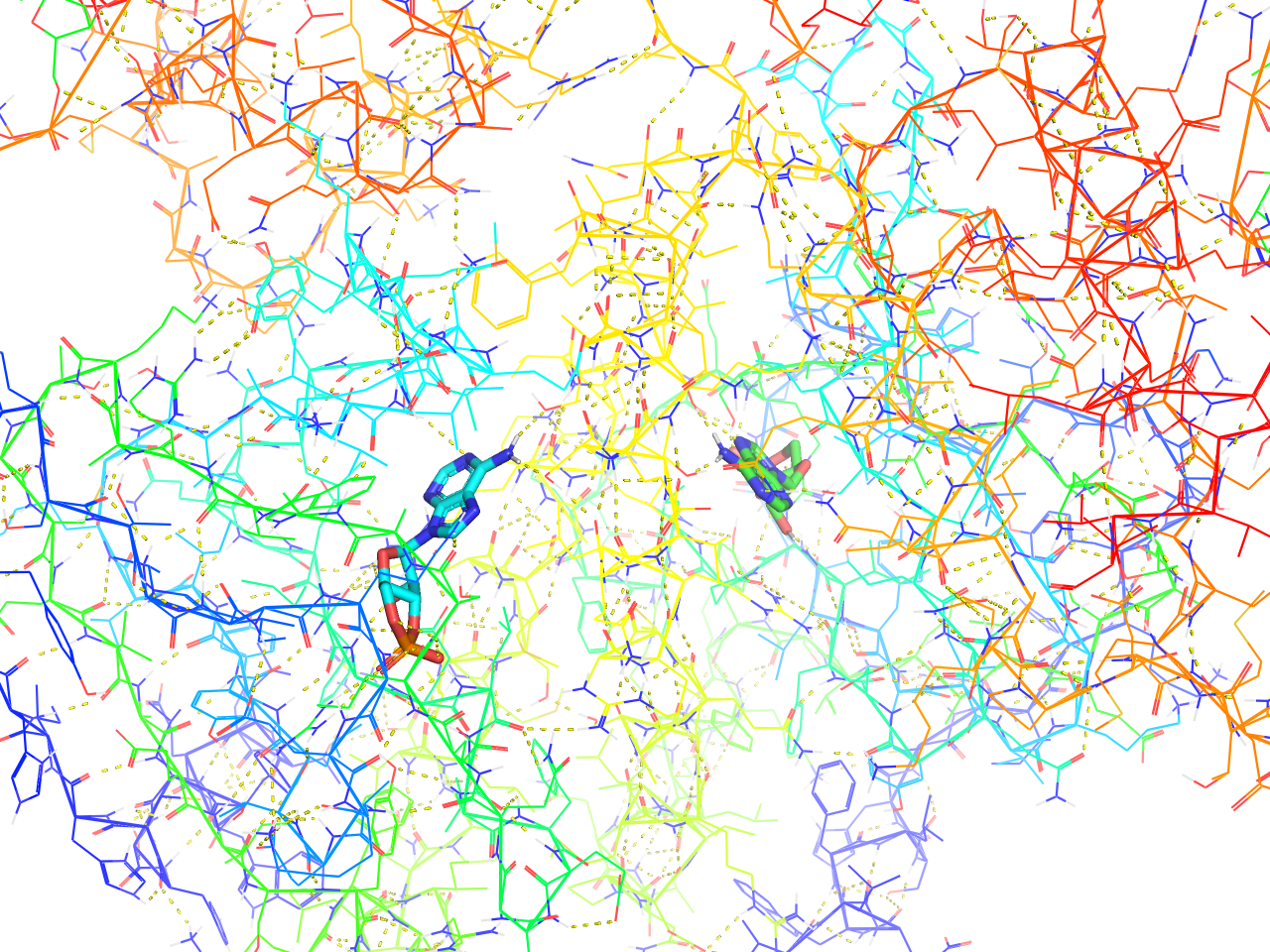
preset.technical(“objectname”,_self=cmd)
ligands
上述のRainbowカラーリングが施され、基本的には主鎖構造のみのribbon表示になりますが、リガンドから一定範囲のみ側鎖を含んだline表示が行われ、リガンドへの水素結合自動検出処理が行われます。
カメラもそのリガンド周辺にズームしてくれますが、リガンドが複数ある場合はそれらの中間にカメラを合わせてしまうようです。
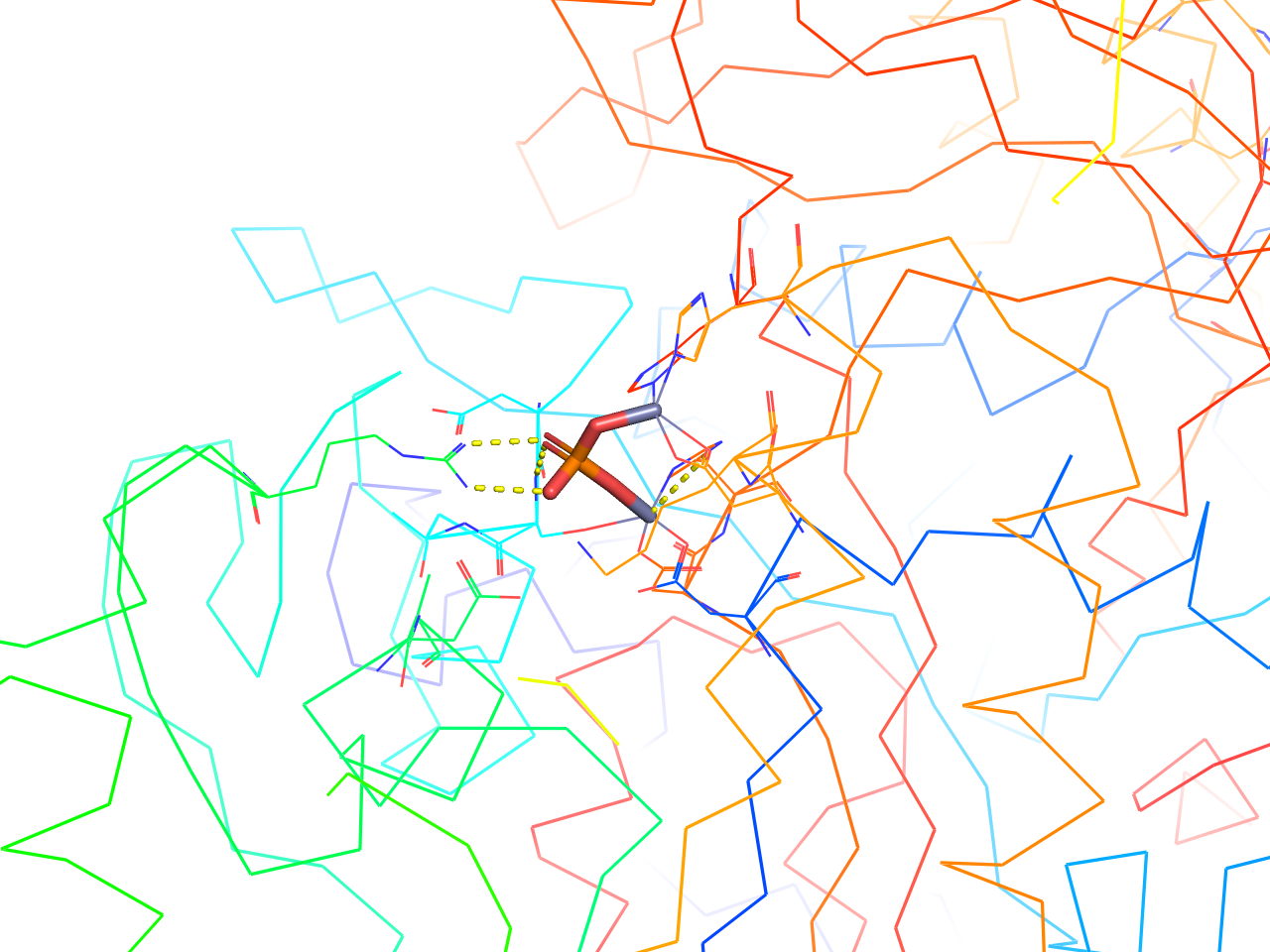
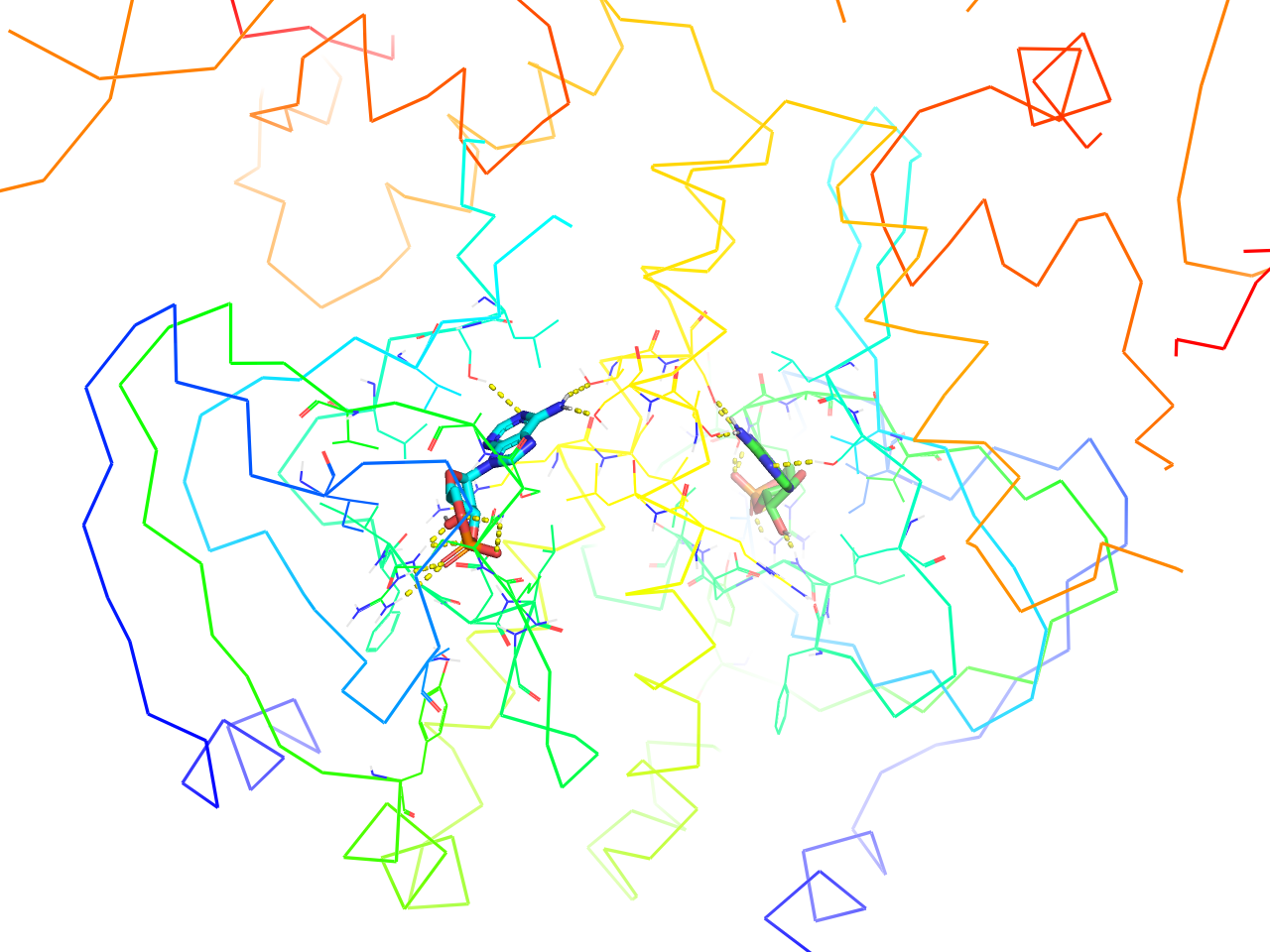
preset.ligands(“objectname”,_self=cmd)
ligand sites
上記ligands設定の拡張版と言えます。様々な表示形式が用意されています。いずれのプリセットでも水素結合を検出し、<objectname>_pol_contsというオブジェクトを生成します。
Cartoon
タンパク質をCartoon表示のままRainbowカラーリング、リガンドをStick表示で、周辺の一定範囲のみline表示にします。
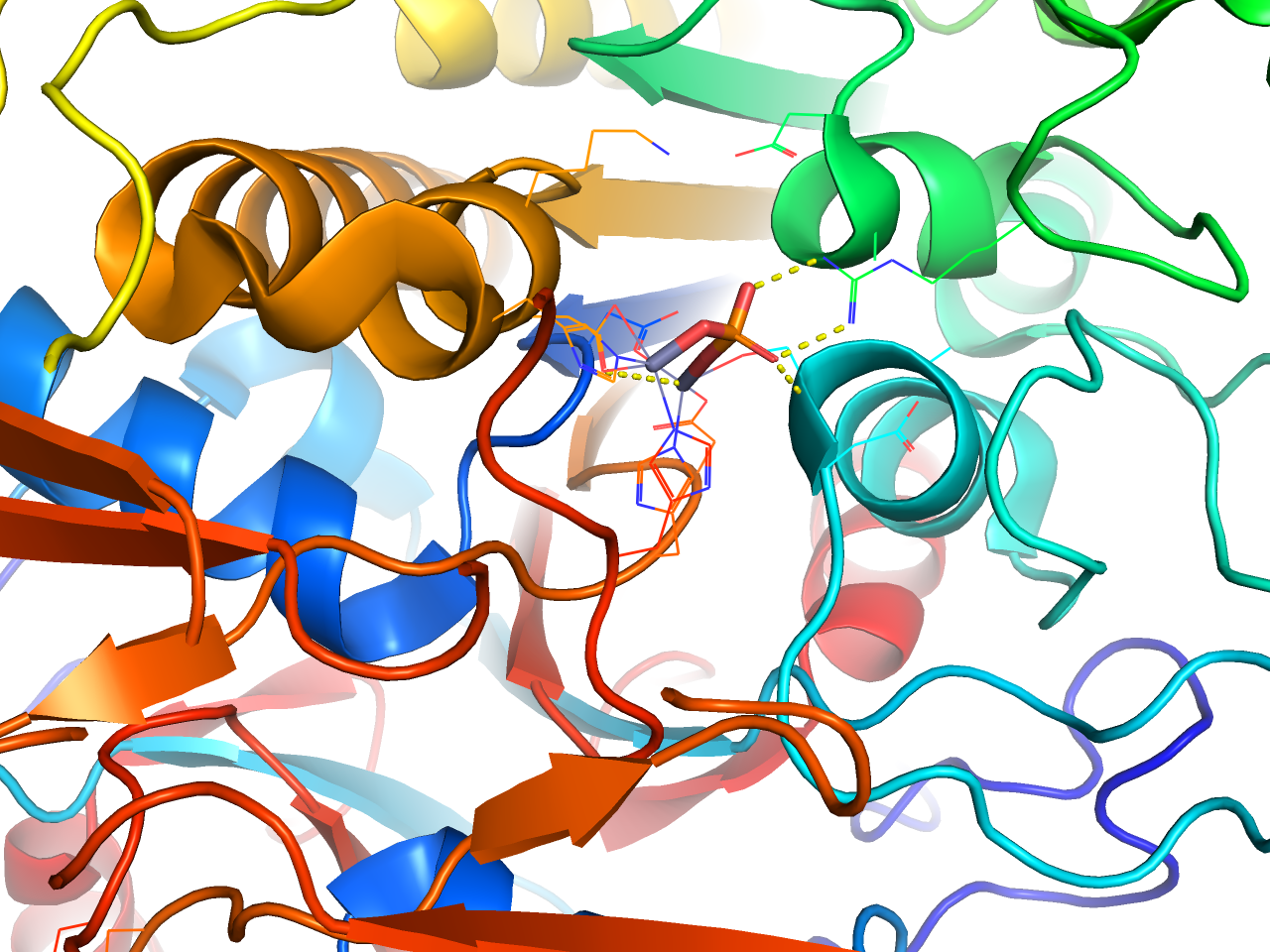
preset.ligand_cartoon(“objectname”,_self=cmd)
solid surface
タンパク質をribbon表示, Rainbowカラーリング, リガンドをStick表示, 周辺の一定範囲のみsurface表示にします。


preset.ligand_sites(“objectname”,_self=cmd)
solid (better)
上記solid surfaceプリセット表示のsurfaceクオリティが上がったものです。設定としてはset surface_quality, 1を追加しています。

preset.ligand_sites_hq(“objectname”,_self=cmd)
transparent surface
上記solid surfaceの透明度を上げ(set transparency, 0.33)、周辺の残基をstick表示にしたものです。
preset.ligand_sites_trans(“objectname”,_self=cmd)
transparent (better)
上記transparent surfaceプリセット表示のsurfaceクオリティが上がったものです。設定としてはset surface_quality, 1を追加しています。

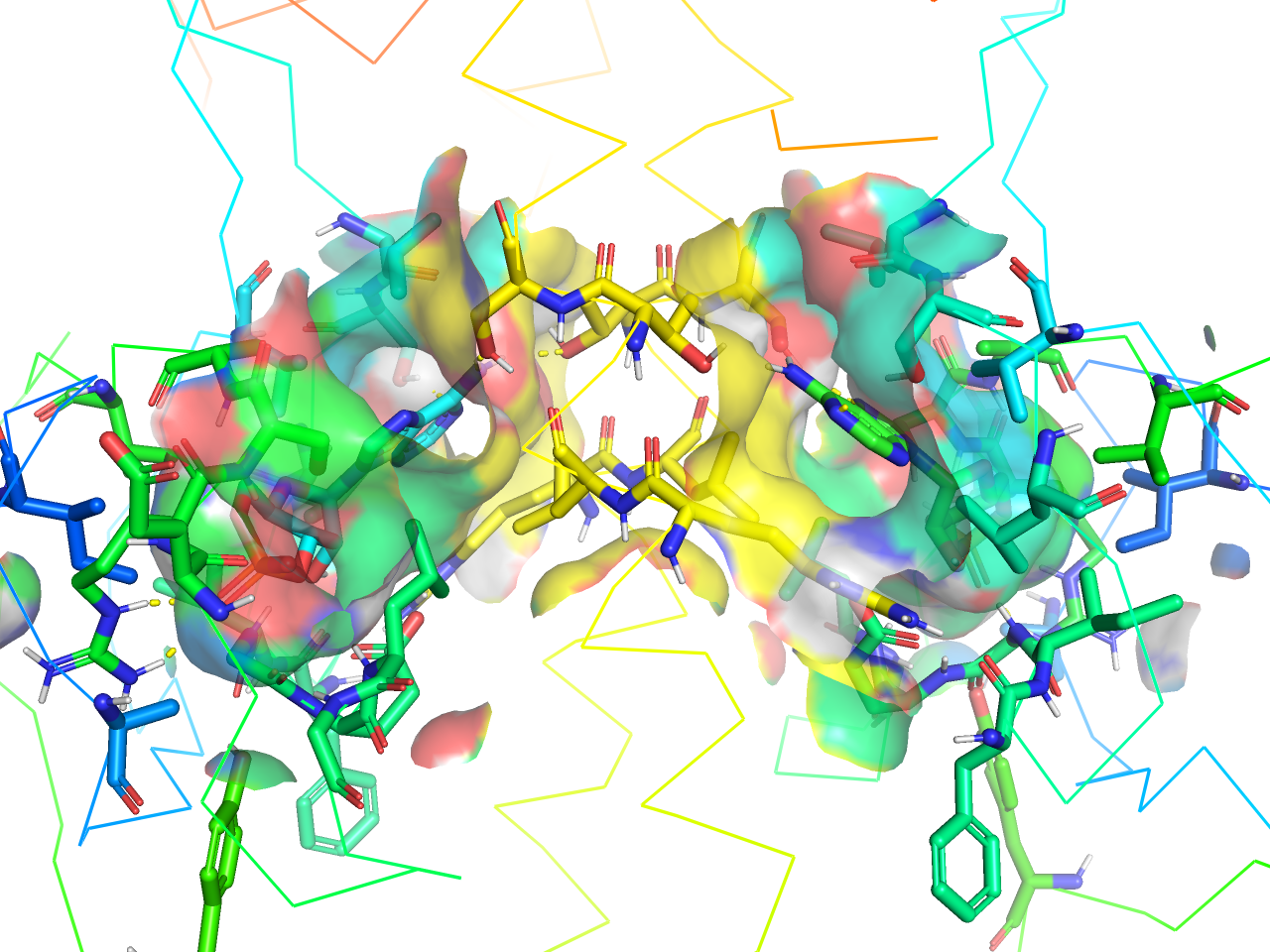
preset.ligand_sites_trans_hq(“objectname”,_self=cmd)
dot surface
上記solid surfaceの表面表示をdotにしたものです。
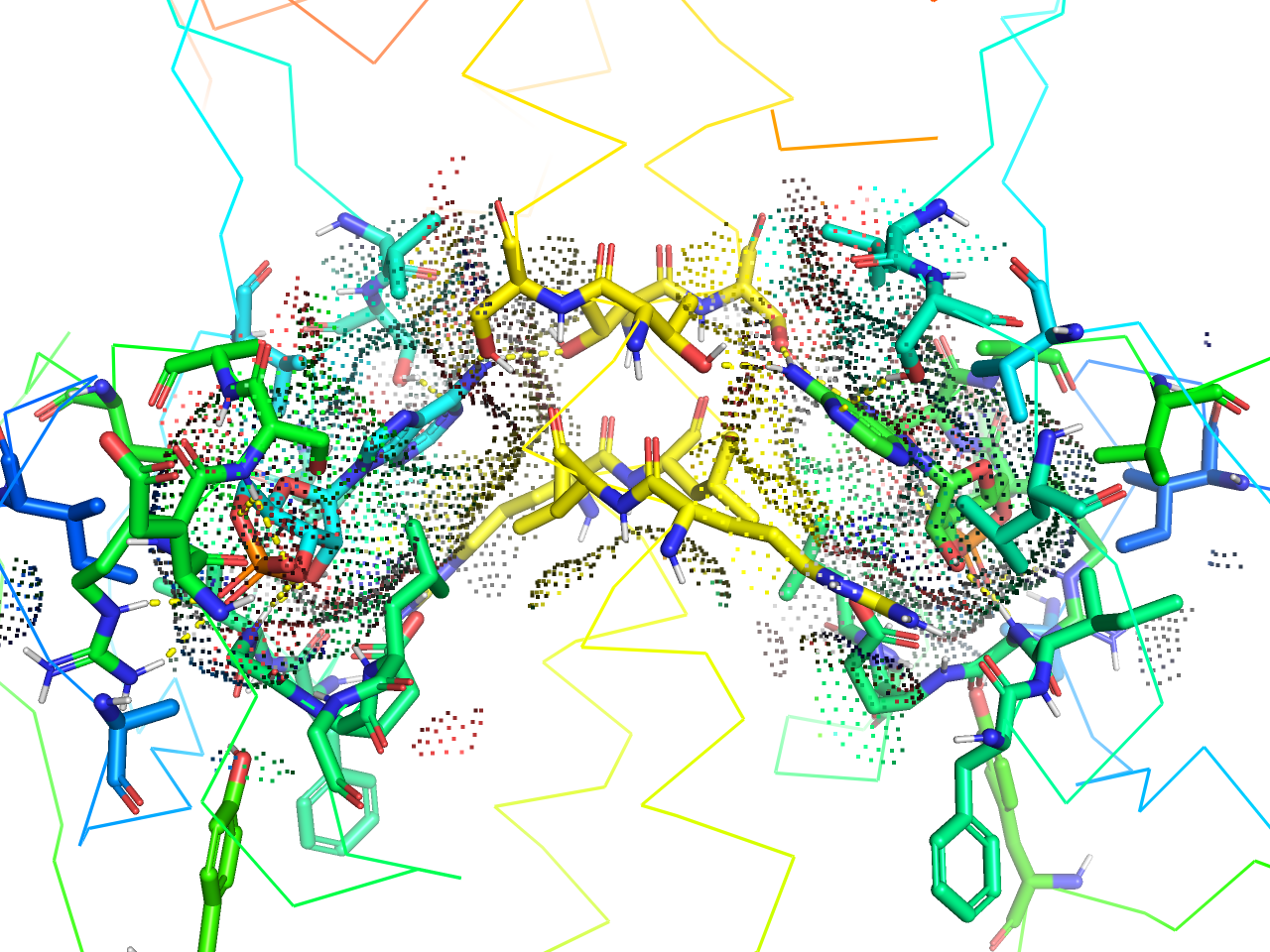
preset.ligand_sites_dots(“objectname”,_self=cmd)
mesh surface
上記solid surfaceの表面表示をdotにしたものです。

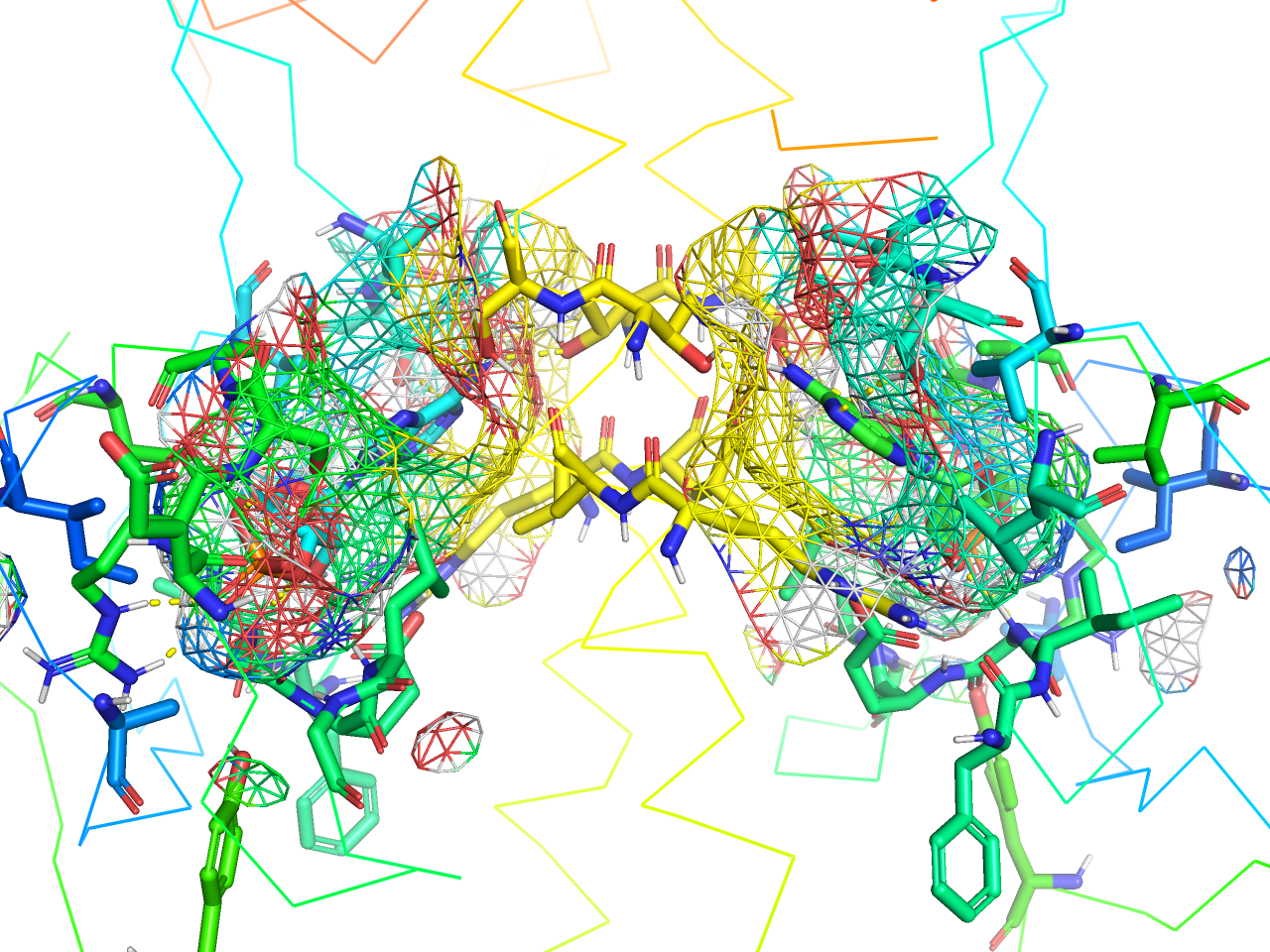
preset.ligand_sites_mesh(“objectname”,_self=cmd)
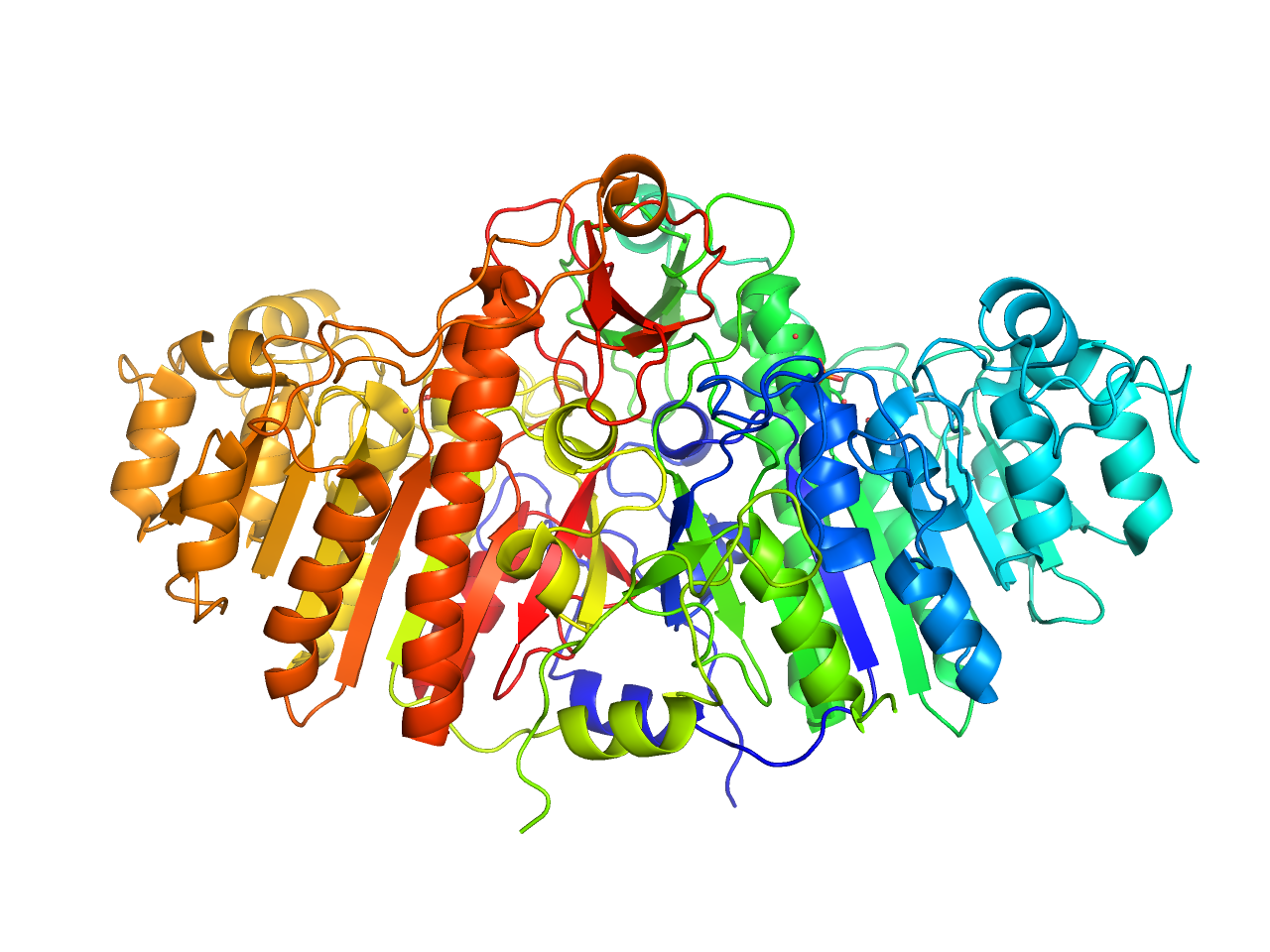
pretty
生体分子のレインボー表示、リガンドをstick形式で表示します。
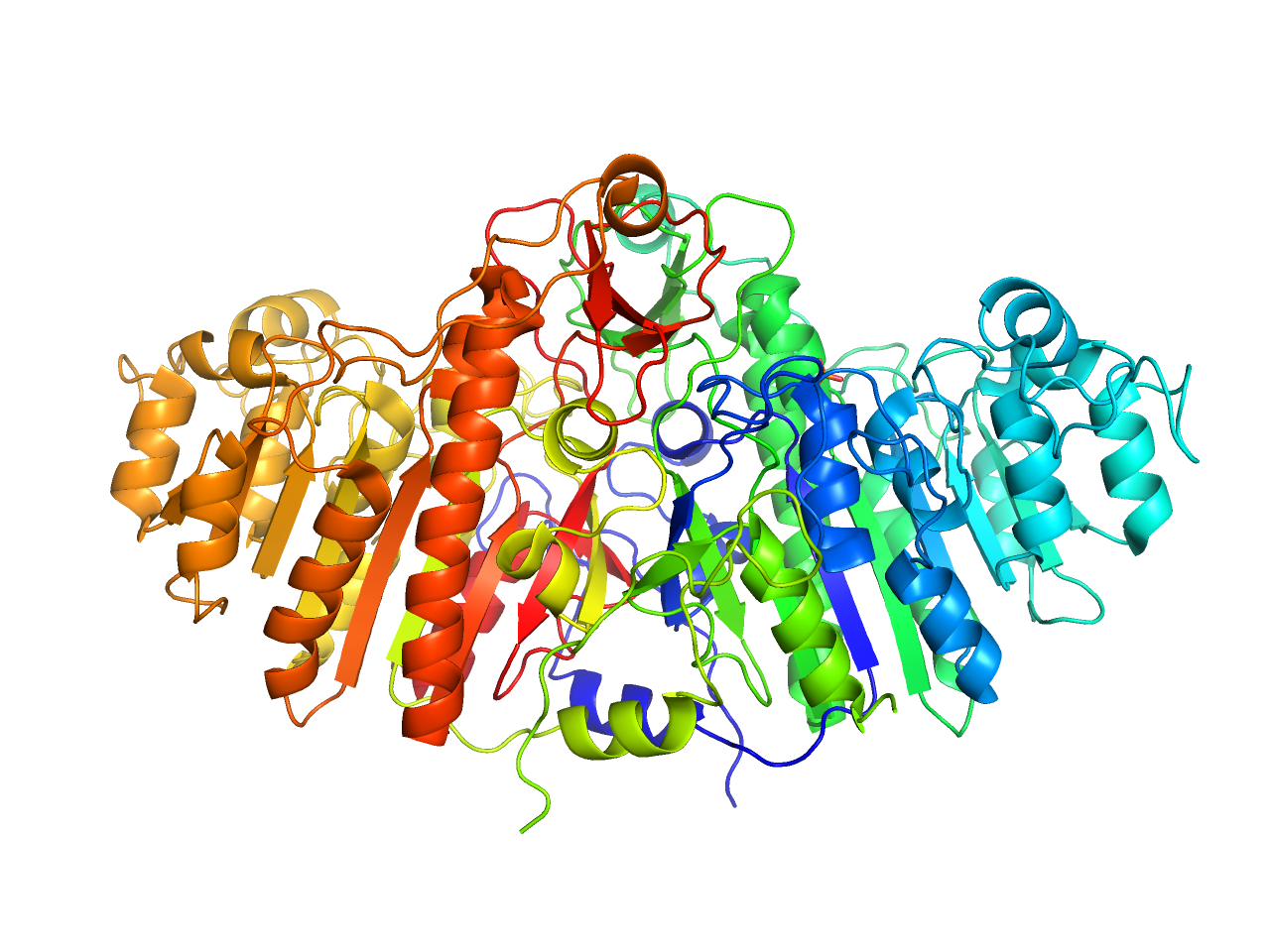
preset.pretty(“objectname”,_self=cmd)
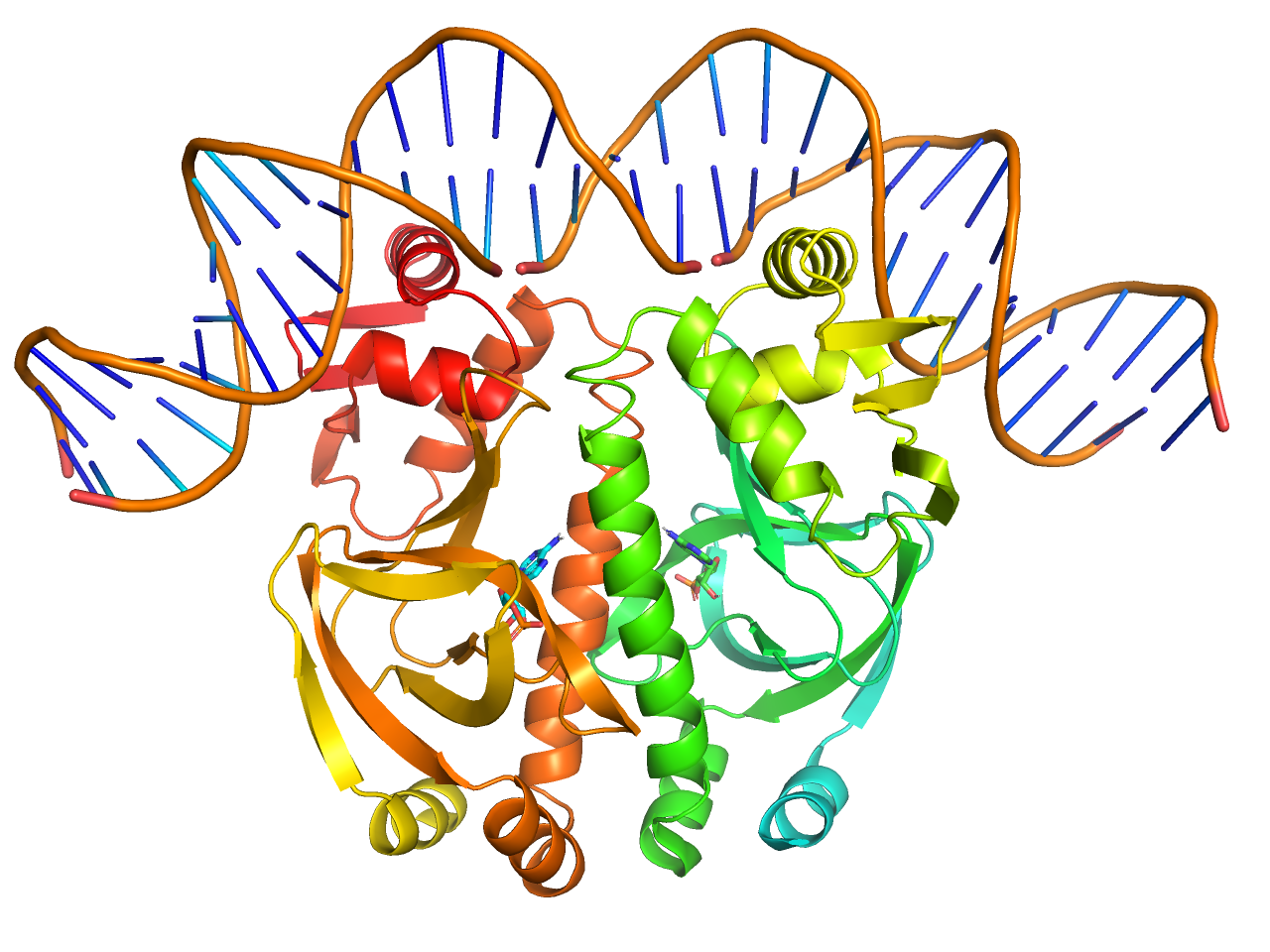
pretty (with solvent)
上記prettyに加えて溶媒やリガンドをnb_spheres表示にします。
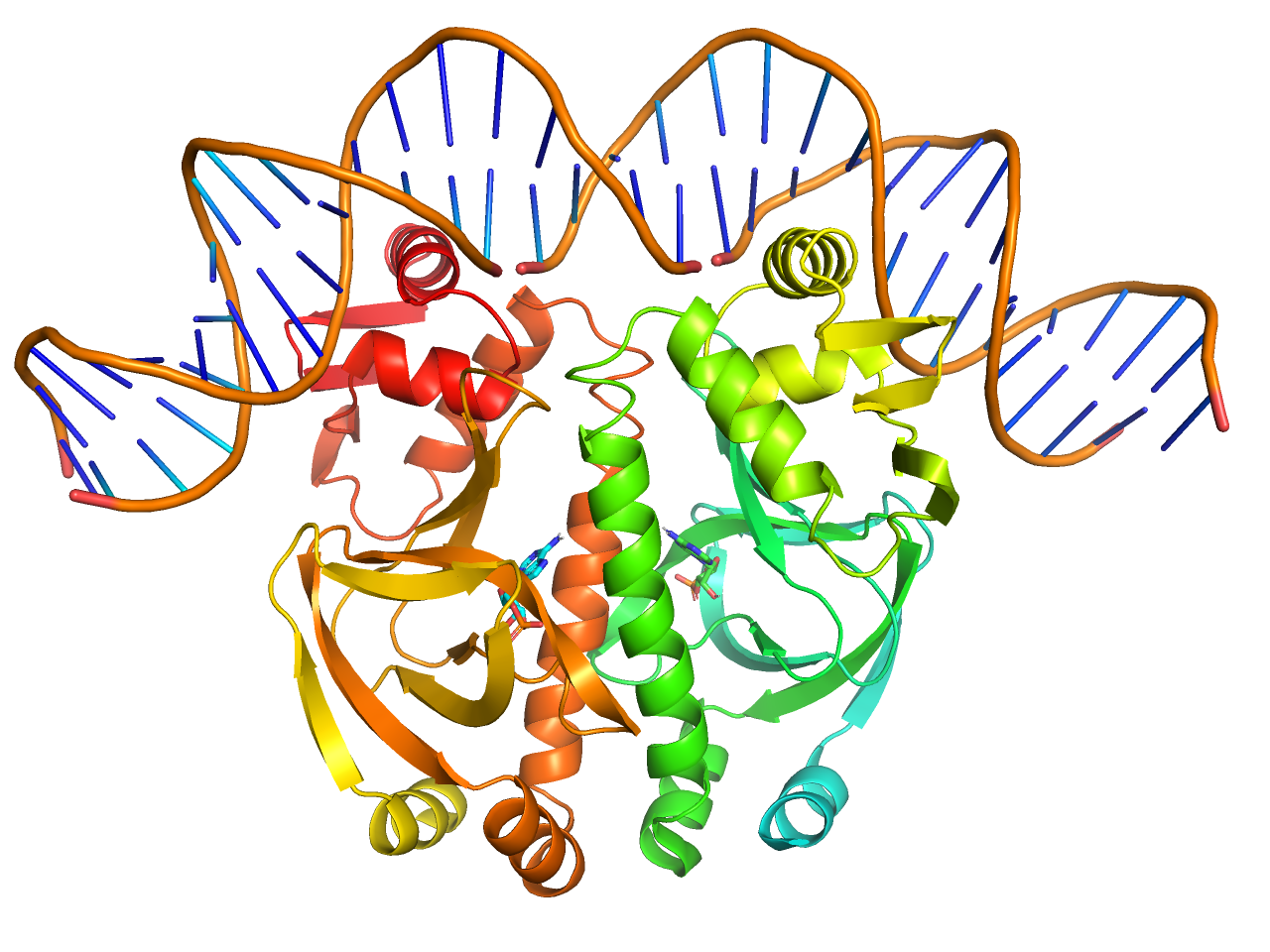
preset.pretty_solv(“objectname”,_self=cmd)
publication
cartoon表示において、
- ループ構造のスムージング
set cartoon_smooth_loops, 1 - ヘリックスやシートの内部を灰色に設定
set cartoon_highlight_color, grey50 - ヘリックスのファンシー化
set cartoon_fancy_helices, 1 - シート構造の平坦化(初期設定でON)
set cartoon_flat_sheets, 1 - 側鎖構造のみの表示(初期設定でON)
set cartoon_side_chain_helper, 0
を行います。
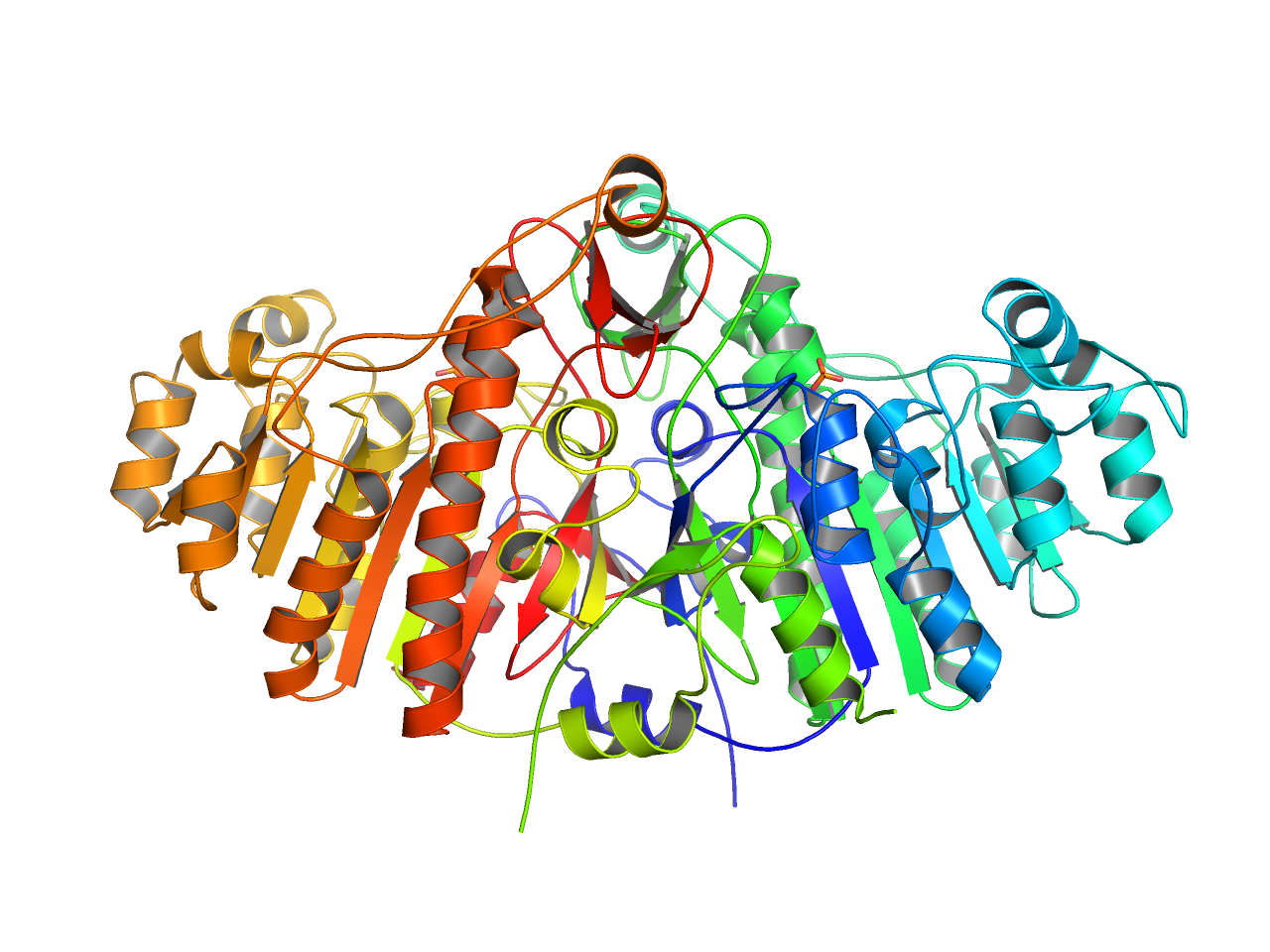
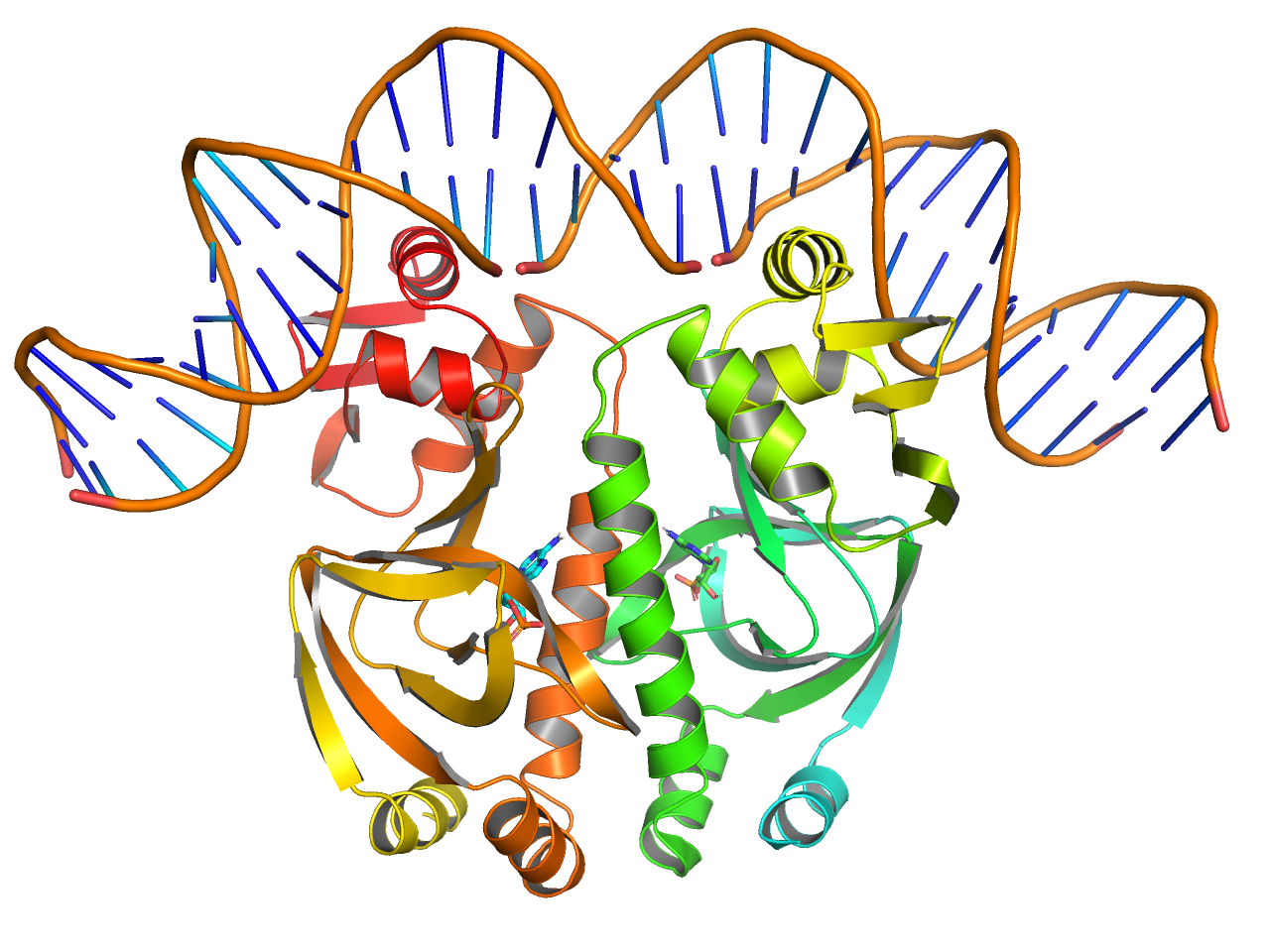
preset.publication(“objectname”,_self=cmd)
publication (with solvent)
上記の溶媒表示版です。
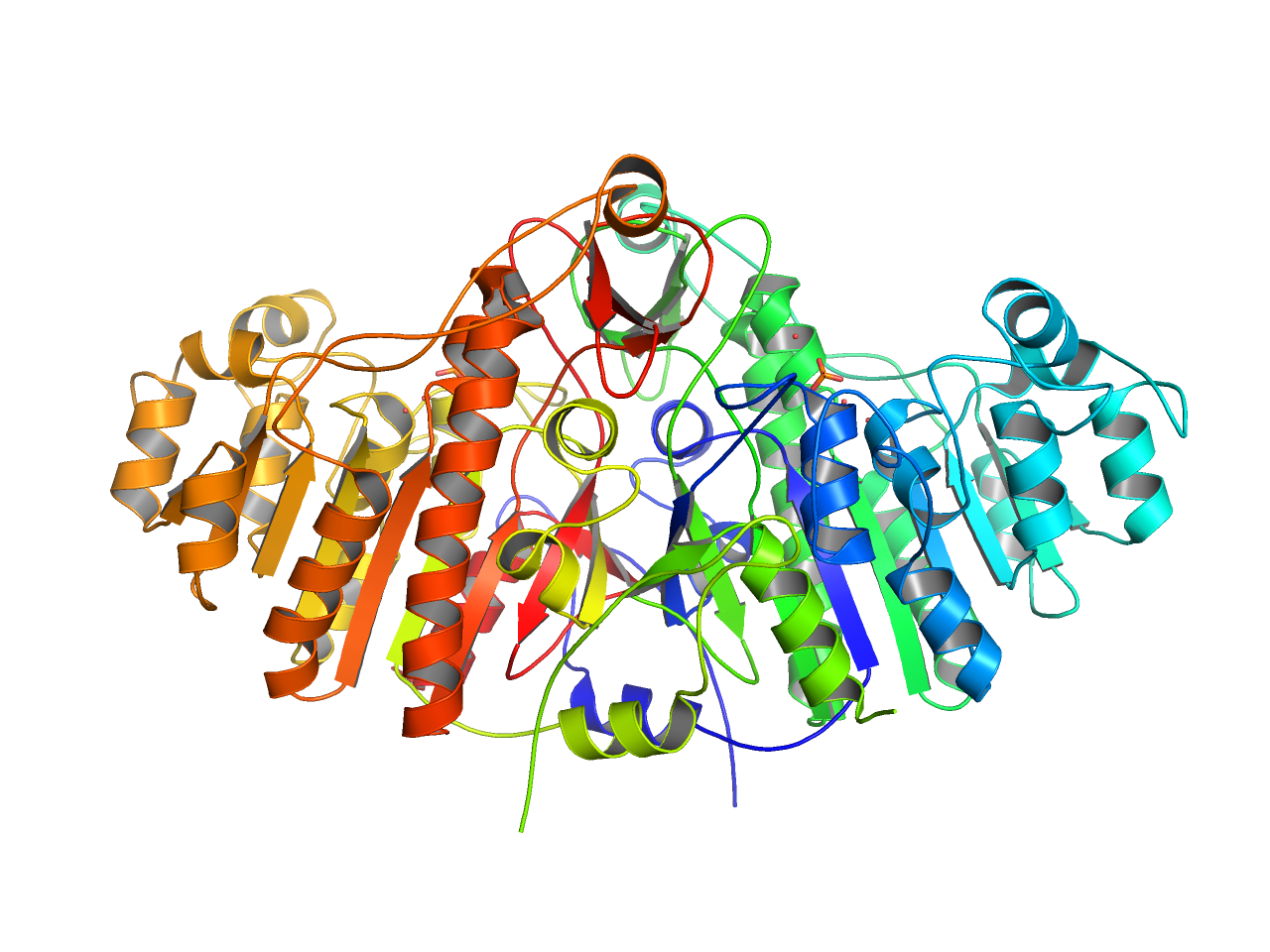
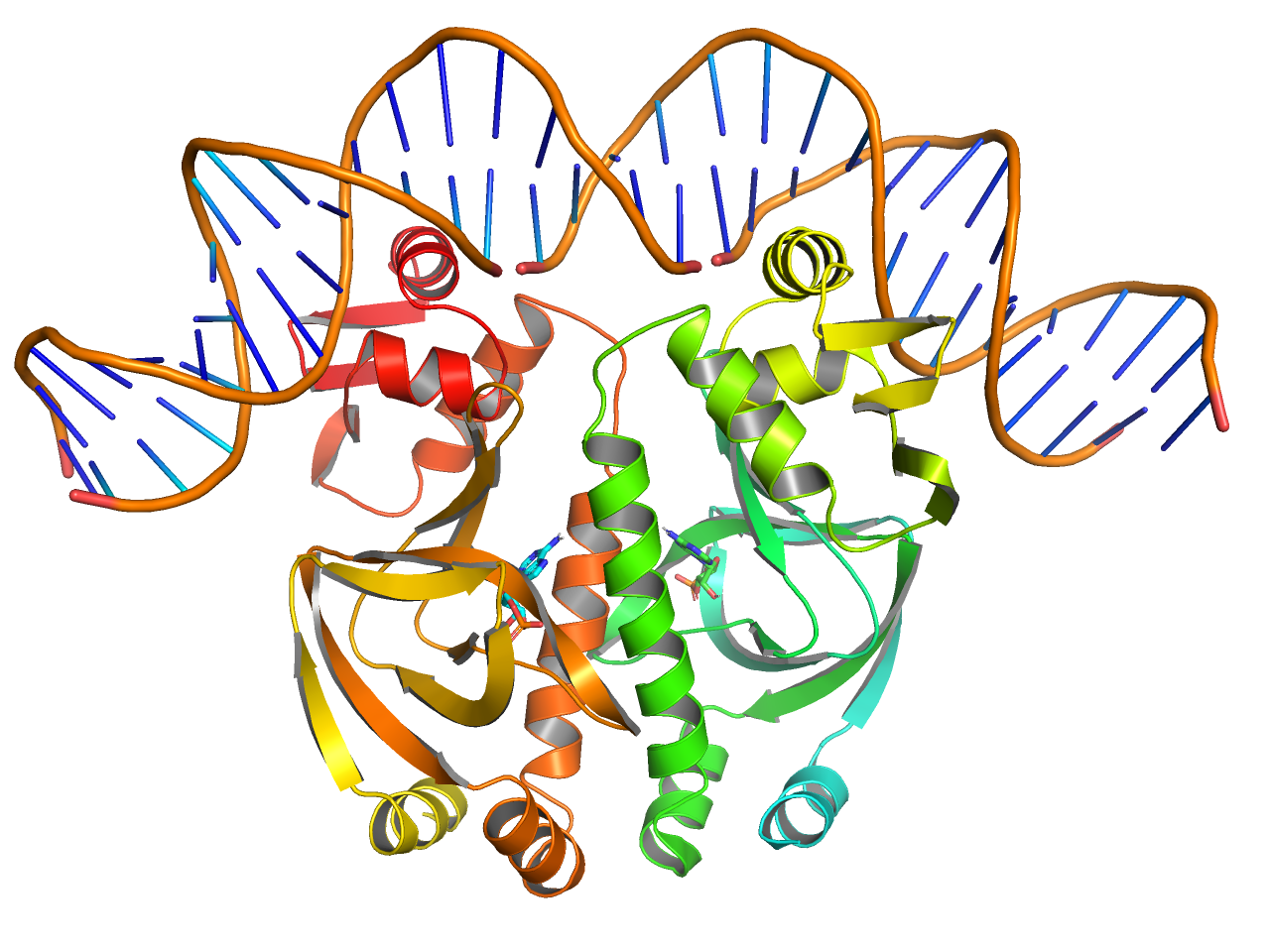
preset.pub_solv(“objectname”,_self=cmd)
protein interface
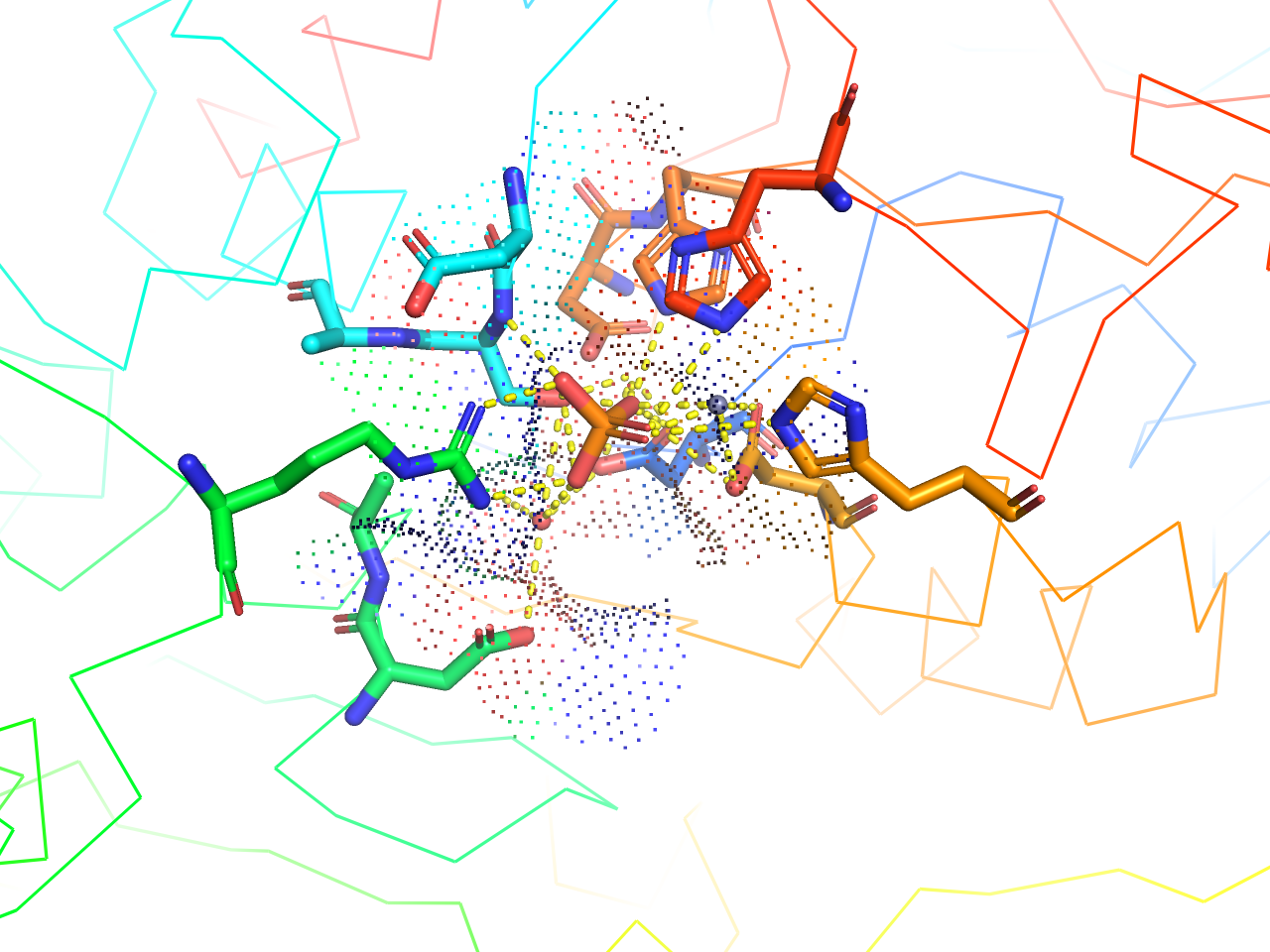
異なるチェインの境目から4.5 Å以内に一部でも含まれる残基をStick表示にします。
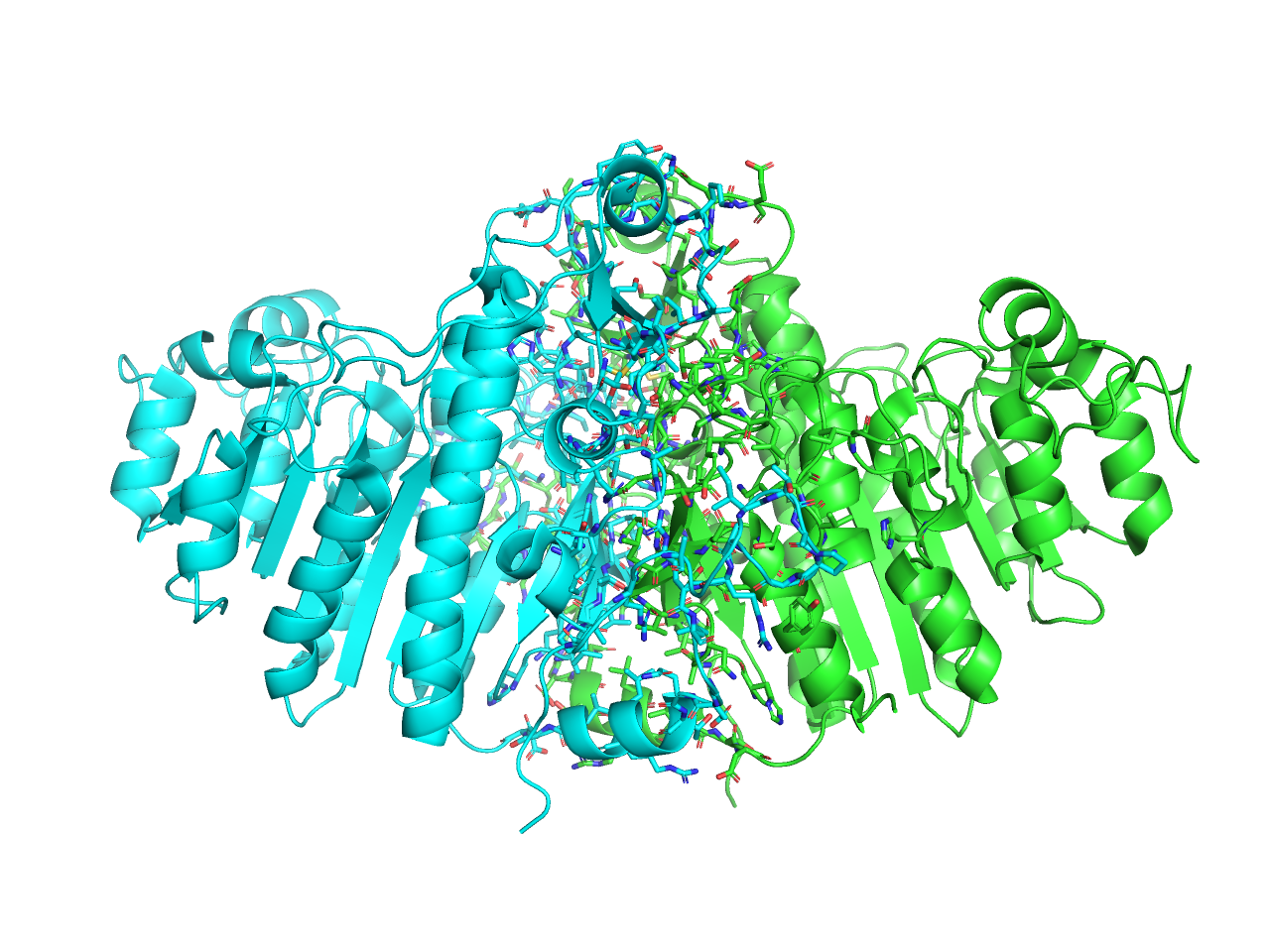
preset.interface(“objectname”,_self=cmd)
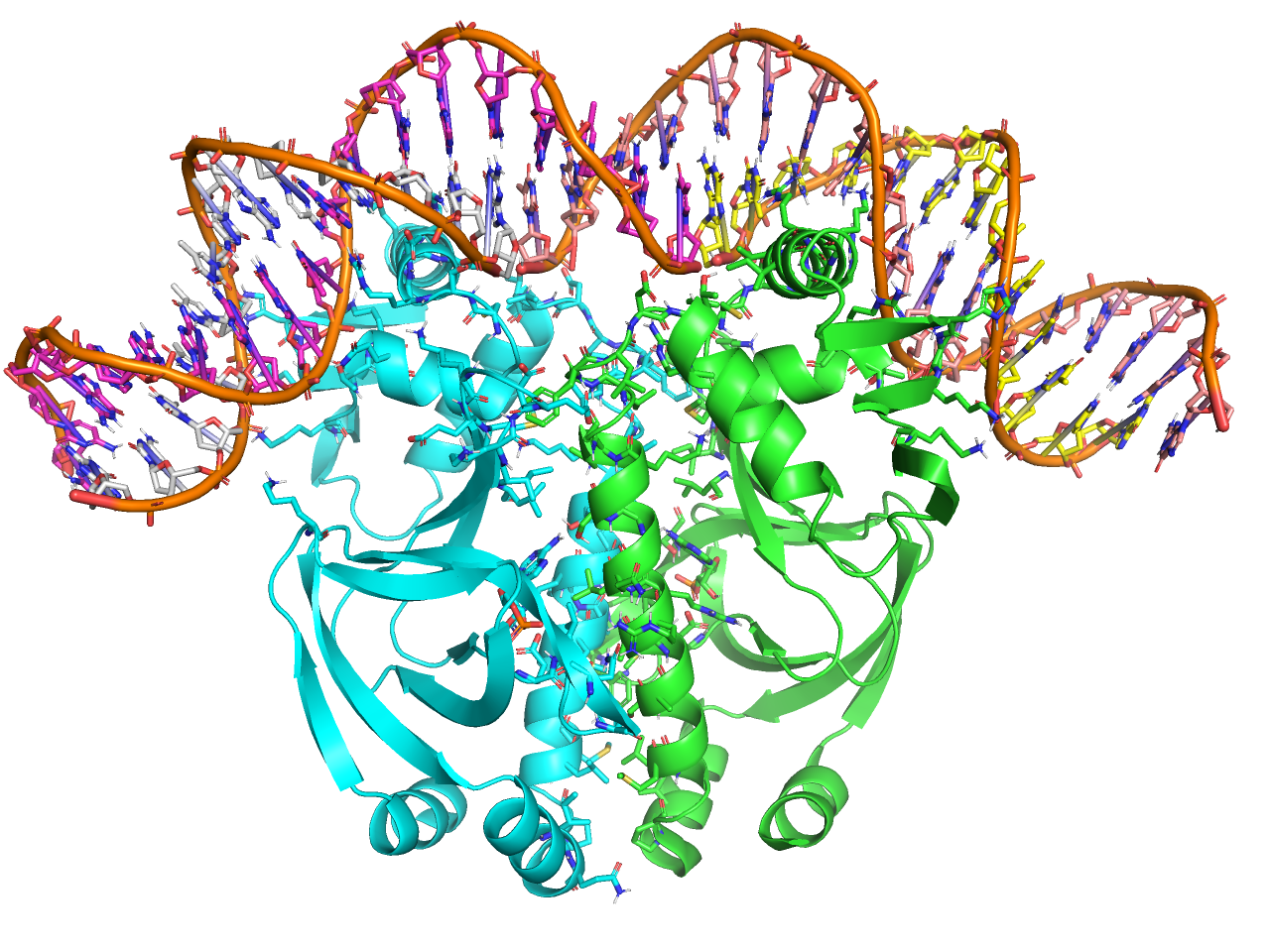
default
PyMOL 1時代はこのシンプルなライン表示だけの形式がデフォルト表示でした。Defaultとついていますが、現在はデフォルト設定ではなくなり、classified presetに取って替わられています。

preset.default(“objectname”,_self=cmd)
Gaussian 16のcubeファイルを開いて分子軌道を表示する
PyMOLは、VMDとまでは行きませんが、様々なファイルの可視化にも対応しています。今回紹介するのは、Gaussian 16などで計算された分子の分子軌道(molecular orbital)をPyMOL上で表示させるテクニックです。
GaussView 6やVMD 1.9.4などの他のソフトを用いて可視化してもよいのですが、タンパク質の構造生物学をやっている人ならだいたい使ったことがあり、扱いに慣れているPyMOLで見られれば、共同研究のときとかに喜ばれると考えられます。
ここでは、簡単な計算の例を示しながら、それをGaussViewまたはPyMOLで分子軌道を表示する時の方法を紹介します。
分子軌道を表示するのに必要な環境
- GaussView 6, VMD, PyMOL 2.3.0以降のうち、いずれか1つをインストールしてあるパソコン
この記事ではGaussView 6またはPyMOL 2.3.0での方法を示します
Example 1-1. シクロペンタジエンの場合
例として、シクロペンタジエン分子の構造をGaussian 16(g16)で構造最適化させ、分子軌道を計算させてみます。インプットファイルcyclopenta.gjf を以下のように書いてg16で計算させます。
%chk=cyclopenta.chk
%mem=20GB
%nprocshared=12
#p opt b3lyp/6-311g(d,p) pop=full
Title
0 1
C -1.78668958 -0.09572728 0.64728372
C -1.36269158 1.27506872 0.64728372
H -1.14231658 -0.96843328 0.64730872
H -0.33812858 1.63156072 0.64725872
C -3.62816658 1.22478072 0.64727472
H -4.64865477 1.54029066 0.71018208
C -3.18843958 -0.09572728 0.64728372
H -3.80595672 -0.96740593 0.58605536
C -2.51974858 2.06652172 0.64717972
H -2.52502738 2.68450968 1.52065726
H -2.55124904 2.68351582 -0.22644835
ここで、重要なのは計算条件を指定する#p opt pop=fullの部分です。opt pop=fullで構造最適化計算と、電子密度解析計算を行うよう指示します。計算レベルや基底関数についてはb3lyp/6-311g(d,p)でなくても好みのものを使ってください。
(ちなみにGaussian 16だと%nprocsharedの指定は非推奨になっています。最近実装された、環境変数での指定方法のほうが使い勝手が良いと思います。さらに、GPUを使った計算をしたい場合にはもっと別の指定方法になります。 参考: http://www.hpc.co.jp/gaussian_Link-0-Equivs.html )
実行コマンド例は
#!/bin/bash
job="cyclopenta"
export GAUSS_CDEF="0-11" # cyclopenta.gjfファイルの%nprocshared=12に対応
g16 < ${job}.gjf > ${job}.log
計算が終わりますと、計算結果のcyclopenta.logファイルだけでなく、.chk(チェックポイント)ファイルcyclopenta.chkも生成されているはずです。分子軌道の可視化や以降の処理にはこのファイルを用います。
Example 1-2. chkファイルの処理
このchkファイルを用いて、まずターミナル上での以下のコマンドで、formatted checkpoint file形式に変換します。
formchk cyclopenta.chk cyclopenta.fchk
として、formatted checkpoint fileに変換します。このformchkコマンドはGaussian 16と同時にインストールされているはずのコマンドです。Gaussian 16がインストールされてあるマシンやスパコンでは、Gaussian 16の本体であるg16コマンドが使えるならば、ほぼ間違いなく使えるはずです。
(ちなみにGaussian 16で計算したchkファイルをGaussian 09時代のformchkコマンドで変換することはできない……かもしれません)
続いて、上のコマンドで作成されたcyclopenta.fchkファイルから、必要な分子軌道のデータを.cube形式のファイルに抽出します。
cubegen 0 MO=homo cyclopenta.fchk cyclopenta_homo.cube
このcubegenについての操作方法はGaussian公式のcubegenの解説ページのページを参照してください。MO=のあとにhomoやlumo、または数字を指定すると、それに対応した分子軌道が出力されます。
今回はシクロペンタジエンのHOMO(最高被占軌道)のデータを取り出したいので、MO=homoとし、.fchkファイルと出力ファイル名cyclopenta_homo.cubeを指定します。
Example 1-3. cubeファイルの表示
Case 1. GaussView 6で表示する
GaussView 6で表示する時、必要になるファイルはcyclopenta.logとcyclopenta_homo.cubeです。まずはふつうにcyclopenta.logを開きます。
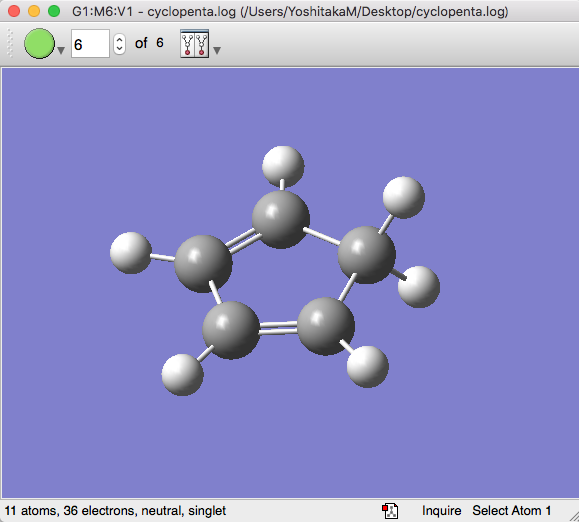
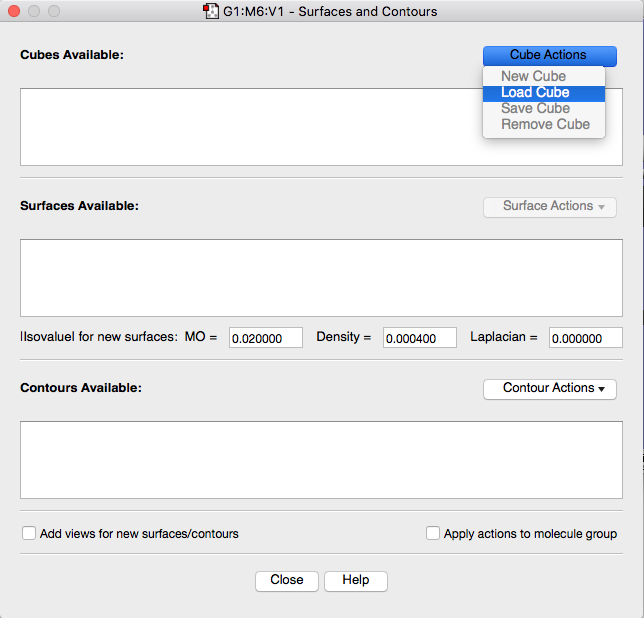
続いて、Results > Surfaces/Contoursを選択し、Cube ActionsからLoad Cubeを選び、cyclopenta_homo.cubeを選択します。
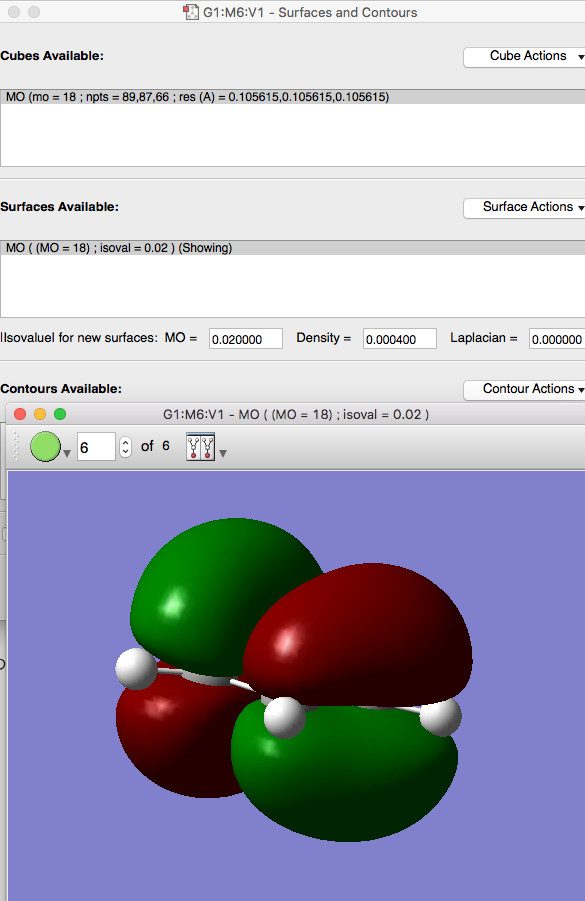
続いて、Surface Actions > New Surfaceをクリックすると、分子軌道の図が表示されるようになります。
このSurfaceの描画モードを変えたい場合は、この紫の画面内で右クリックし、View -> Display Formatを選択します(macOSはCommand+DでもOK)。
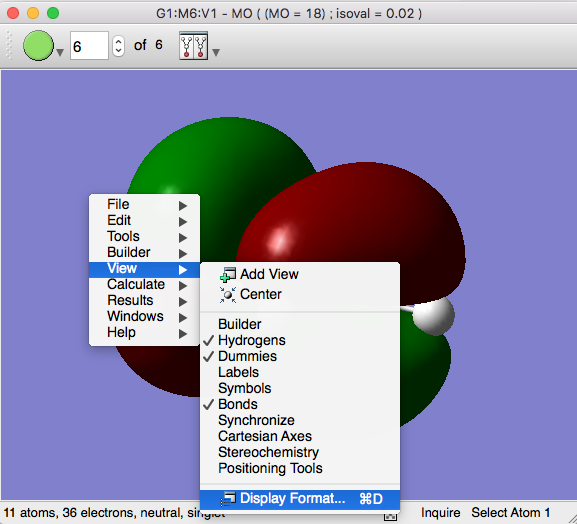
このDisplay Formatウィンドウの中で、右端のSurfaceタブを選び、Format: transparentとすれば、半透明な表面を描画することができます。透明度はそこのスライダで調節できます。またmesh表示も可能です。
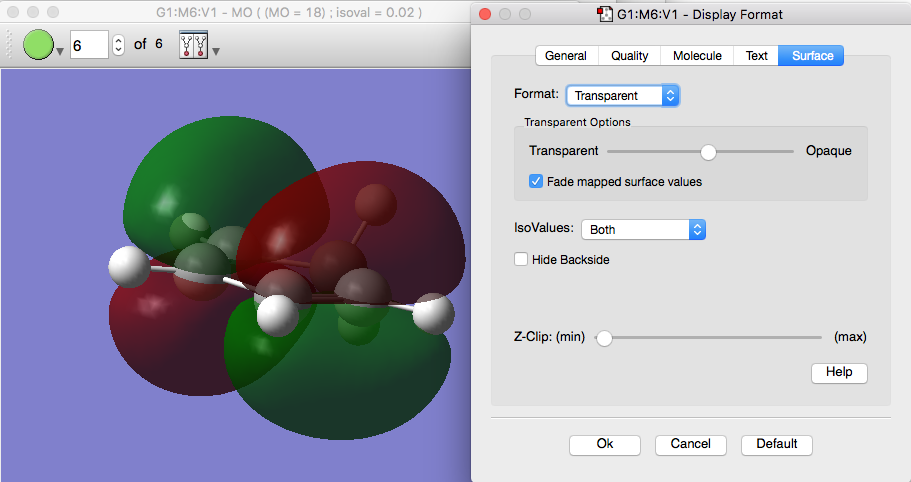
以下では、この部分をPyMOLでやってみる方法を紹介します。
Case 2. PyMOL 2.3.0で表示する
PyMOLで開く場合には、
cyclopenta.logの最終構造に対応する構造ファイルをPDB形式などで用意し、PyMOLに表示させる- この上に
cyclopenta_homo.cubeをロードし、適切な処理を施す。
という流れになります。このうち、1.で述べた構造ファイルを用意する部分はやや面倒かもしれません。お使いのマシンにAmberTools 18がインストールされている状態であれば、
antechamber -i cyclopenta.log -fi gout -o cyclopenta.pdb -fo pdb
とすることで最終構造のPDBファイルを一発変換できます。AmberToolsがない場合は、オープンソースのファイルコンバータであるOpen Babelを使った変換法で代用できます。Homebrewのインストール方法は適当にググってください。
# Open BabelをHomebrewでインストール
brew install open-babel
# Usage:
# obabel [-i<input-type>] <infilename> [-o<output-type>] -O<outfilename> [Options]
# input-typeにはまだg16フォーマットがサポートされていないのですが、g09で代用可能だと思います。
# see also 'http://openbabel.org/docs/current/FileFormats/Overview.html#file-formats'
obabel -i g09 cyclopenta.log -o pdb -O cyclopenta.pdb
こうしてファイル形式を変換して作成したcyclopenta.pdbをPyMOLで開いてみます。
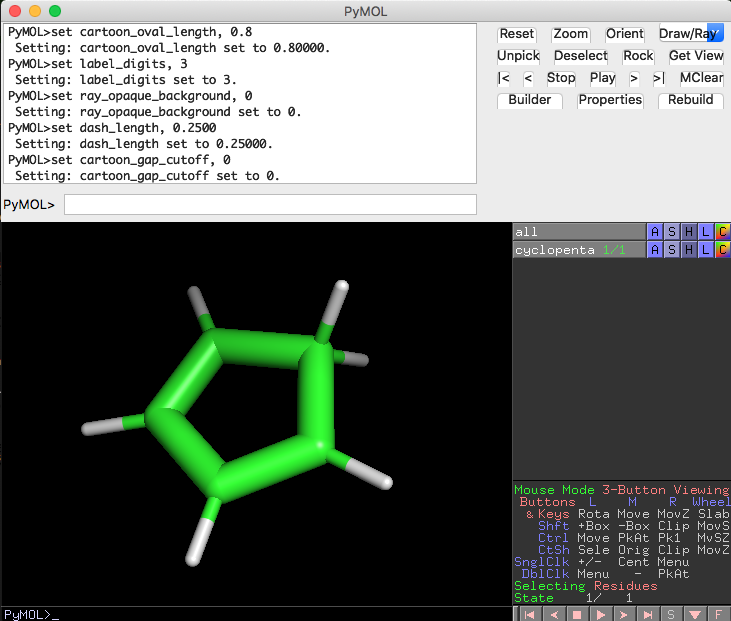
んー、本来は二重結合になっている炭素の結合情報が、全部同じような線で繋がれてしまっていますね。これが嫌だな〜って方は、以下のようにして二重結合っぽい表示に変えてみます。
二重結合にしたい原子の上でそれぞれ右のダブルクリック(マウスにホイールがある場合はホイールクリックでも可能)をすると、Pk1, Pk2という選択印が付きます。この状態で、PyMOLのコマンド unbond ; bond order=2 を実行します(コマンド入力できるフォームは2箇所ありますが、どちらに入れても同じです)。このorder=2で結合次数を指定しています。
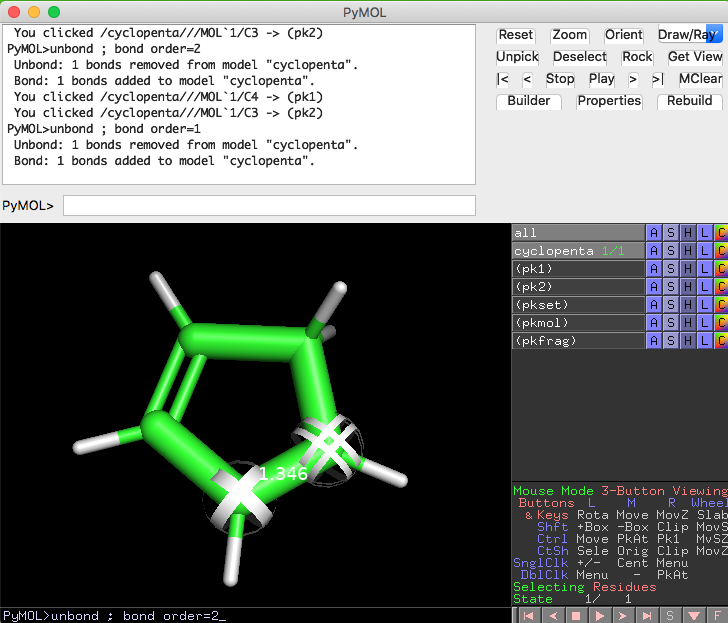
これで二重結合っぽい表示に変わりました。ついでに、好みで以下の設定を入れてGaussViewっぽい描画設定にしてみます。
show sticks
show spheres
set stick_radius, 0.1
set sphere_scale, .22
set sphere_scale, .18, elem H
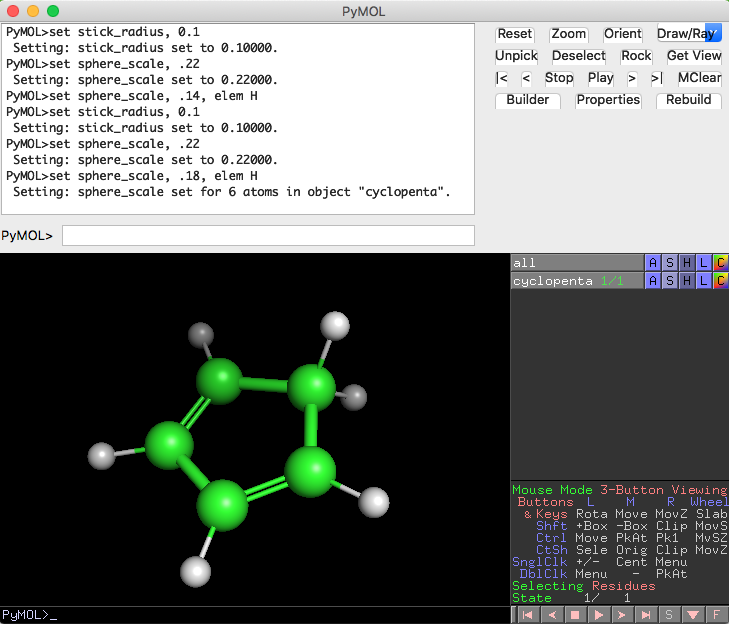
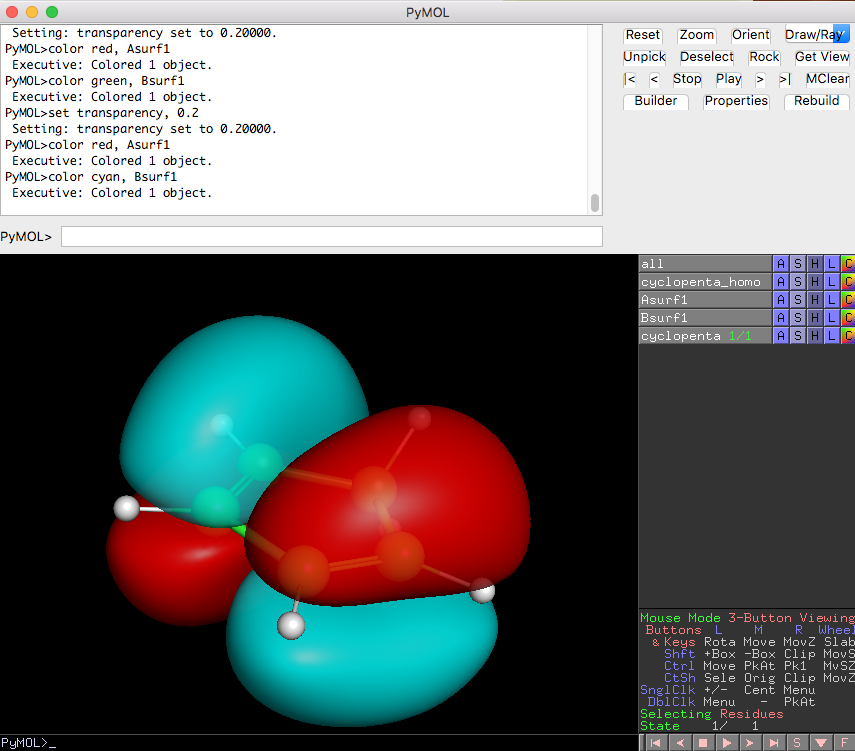
では、PyMOLにcyclopenta_homo.cubeファイルをロードします。コマンドは以下の通り
load /path/to/cyclopenta_homo.cube
isosurface Asurf1, cyclopenta_homo, 0.02
isosurface Bsurf1, cyclopenta_homo, -0.02
color red, Asurf1
color blue, Bsurf1
set transparency, 0.5
load部分は、cyclopenta_homo.cubeのファイルのあるファイルパスを指定します。デスクトップ上に置いてあるならばload ~/Desktop/cyclopenta_homo.cubeみたいに。以下のisosurfaceコマンドで分子軌道をしきい値0.02, -0.02で作成します。正と負の波動関数に対応する各電子雲をAsurf1, Bsurf1というオブジェクト名で作成し、色付けを red, blueにしています。
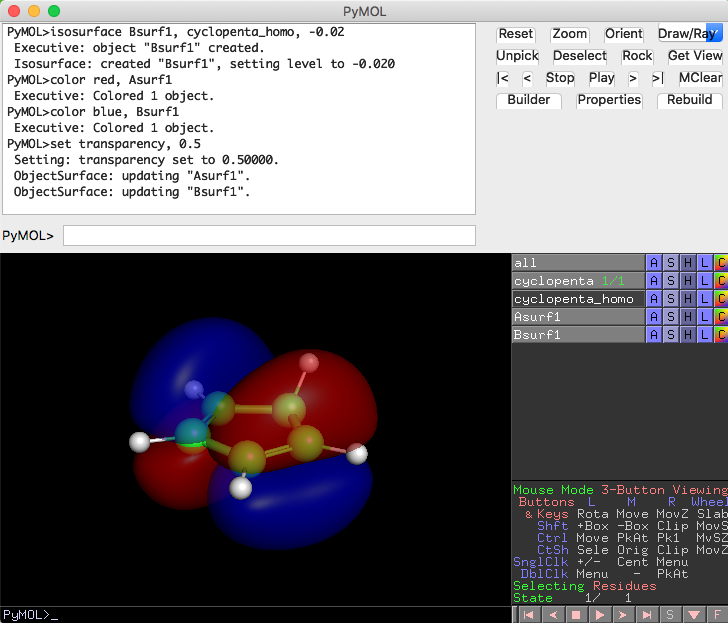
Asurf1, Bsurf1の色変更は、PyMOLのオブジェクト色変更と同じ感覚でマウスを使って簡単に変更できます。
PyMOL上でのpythonスクリプトの実行:基本編
PyMOLの大きな強みの1つとして、PyMOLのコマンドラインからpythonスクリプトを実行させることができることが挙げられます。ここではいくつかの例を挙げながら、PyMOL上でのpythonスクリプト実行機能を紹介してみます。ただし、python3についての基本的な知識があることを前提とします。
Pythonの設定を確認する
まずは現在PyMOLが動作しているPython環境を確認するために、バージョン情報とPATHをPyMOLのコマンド入力欄から確認してみましょう。コマンドは通常のpythonと同じように
# pythonのバージョンを表示
import sys
print(sys.version)
# pythonのモジュール検索PATHを確認
print(sys.path)
となります。返り値は、私の環境(macOSのHomebrewでインストールした場合)では
# PyMOL
PyMOL>import sys
PyMOL>print(sys.version)
3.14.0 (main, Oct 7 2025, 09:34:52) [Clang 17.0.0 (clang-1700.3.19.1)]
# PyMOL>print(sys.path)
['/opt/homebrew/Cellar/pymol/3.1.0_2/libexec/bin', '/Users/YoshitakaM/apps/ambertools25/lib/python3.12/site-packages', '/opt/homebrew/Cellar/python@3.14/3.14.0_1/Frameworks/Python.framework/Versions/3.14/lib/python314.zip', '/opt/homebrew/Cellar/python@3.14/3.14.0_1/Frameworks/Python.framework/Versions/3.14/lib/python3.14', '/opt/homebrew/Cellar/python@3.14/3.14.0_1/Frameworks/Python.framework/Versions/3.14/lib/python3.14/lib-dynload', '/opt/homebrew/Cellar/pymol/3.1.0_2/libexec/lib/python3.14/site-packages', '/opt/homebrew/opt/numpy/lib/python3.14/site-packages', '/opt/homebrew/opt/pyqt/lib/python3.14/site-packages', '/opt/homebrew/lib/python3.14/site-packages', '/opt/homebrew/lib/python3.14/site-packages/coot', '/opt/homebrew/Cellar/modeller/10.8/modlib', '/Users/YoshitakaM/Desktop/work/pymol-psico']
のようになりました。print(sys.path)で表示されたPATHからはモジュールをimportすることができます。
PythonスクリプトをPyMOL上で実行する
PyMOLコマンドラインからはpythonとpython endという入力の間に任意のpythonスクリプトを挟むことで、PyMOL上で擬似インタラクティブにコマンドを実行することができます。ただし、一度pythonを入力した後はpython endを入力するまではフィードバックが得られないことに注意しましょう。
例えば、以下のように変数xに10という値を入れてそれをprintさせるだけの簡単なスクリプトをコマンドラインに入力してみます。
python
x = 10
print(x)
python end
PyMOLのアウトプットとしては
PyMOL>python
PyMOL>x = 10
1:x = 10
PyMOL>print(x)
2:print(x)
PyMOL>python end
PyMOL>python end
10
というように表示され、最後に10という結果がprintされたことがわかります。
この機能を使えば、Pythonを使い慣れた方であれば様々な応用可能性があることに気づくと思います。例えば、あるディレクトリの中で目的の構造ファイル群だけPyMOL上にロードしたいという例では、以下のようにPythonスクリプトを書くことができます。
# globモジュールをインポートし、ワイルドカード*によって
# 拡張子がcifであるファイルを一括でPyMOL上にロードする
python
from glob import glob
for file in glob("*.cif"):
cmd.load(file)
python end
コマンドを外部ファイルに保存し、PyMOLからスクリプトを呼び出す
上で挙げた一括ロードのPythonスクリプトを繰り返し使いたいときは、別ファイルにスクリプトを保存しておいてそれを呼び出すような形にすれば、毎回入力しなくて済むようになります。この場合は、pythonとpython endの間の部分だけを別のファイル(名前はcifload.pyとします)に書いておきます。
from glob import glob
for file in glob("*.cif"):
cmd.load(file)
これをPyMOL上から呼び出すときには、コマンドラインからrun /path/to/cifload.pyとして呼び出します(/path/to/の部分はcifload.pyが存在するディレクトリパスに適宜置き換えてください)。
拡張コマンドを使えるように読み込む
発展的な内容ですが、上記の方法を使えばPyMOLWikiのScript Libraryなどで公開されている拡張コマンドを即座に使えるようにすることもできます。例として, タンパク質の色分けをアミノ酸の疎水性〜親水性に応じて行うcolor_h, color_h2コマンド(https://pymolwiki.org/index.php/Color_h)を使えるようにします。
上記ページのコードのfrom pymol import cmdからcmd.extend('color_h2',color_h2)の前にpythonを、最後にpython endを入力することで、color_h, color_h2コマンドが使えるようになります。
これによってcolor_h, color_h2の拡張コマンドが使えるようになりました。もちろん、外部ファイルに保存しておいてrun ~~で呼び出すことも可能です。