アルカリホスファターゼの加水分解酵素としての働き
最後に、アルカリホスファターゼが「ホスファターゼ」の名前通りにリン酸モノエステル加水分解酵素として化学的にどう働いているかを観察してみましょう。
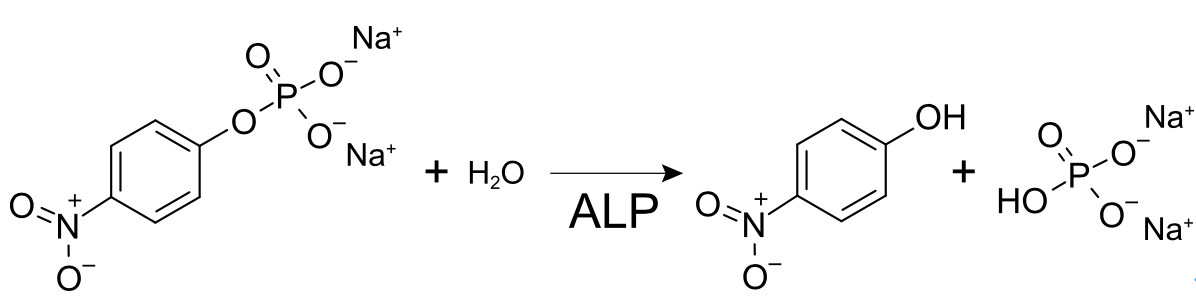
アルカリホスファターゼ(ALP)は以下の反応式のように、p-ニトロフェニルリン酸を加水分解してp-ニトロフェノール(黄色く発色する)を生成することができます。
この反応の様子をPyMOLで見てみましょう。
このリンクに1alk_reaction.pseという名前のPyMOLセッションファイルを置いています。これをダウンロードしてダブルクリック、またはFile -> Openからこのファイルを開いてみましょう。このセッションファイルにはアルカリホスファターゼの単量体とともに、今まで無機リン酸が入っていた箇所にp-ニトロフェニルリン酸(残基名:NPH)の構造が見えるはずです。
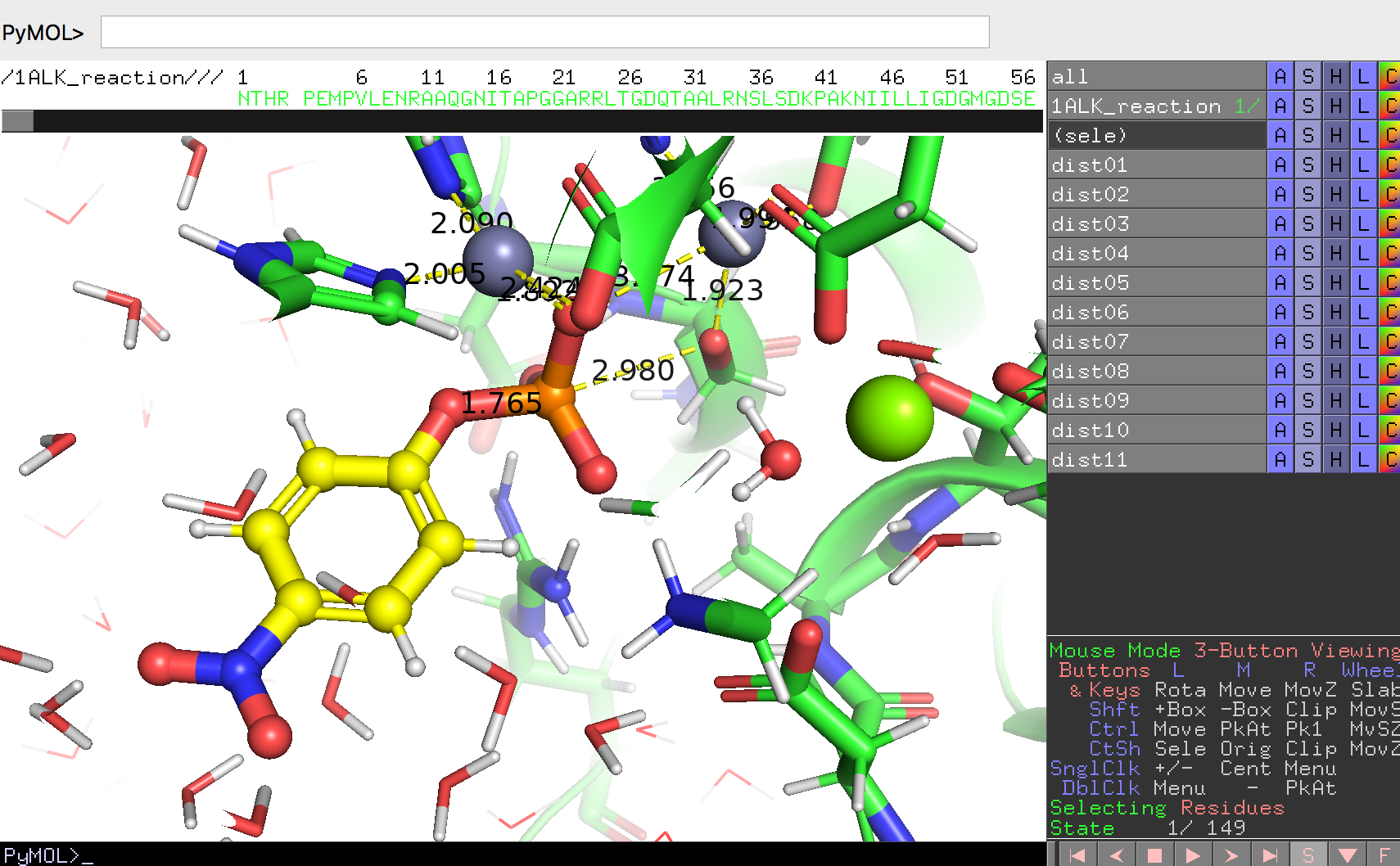
PyMOL Internal GUIの右下には▶ボタンがあります。この▶ボタンを押すとタンパク質とp-ニトロフェニルリン酸が少しずつ動き、化学反応を起こしている様子が可視化されて見えるはずです。
このような動いている様子が見られるのは、このセッションファイルにはあらかじめ149フレーム分の構造データを入れたオブジェクトが含まれているからです。ほとんどすべてのRCSB PDBからダウンロードしてきた結晶構造のデータは静止しているために1フレーム分のデータしかありませんが、NMRで構造決定されたデータですと、タンパク質の「動き」が収録されているものもあります(例としてPDB ID: 1G03)。
このセッションファイルには、dist01からdist11までの距離オブジェクトも含まれています。この距離オブジェクトはマウス操作の原子ピッキング(PkAt)機能を用いることで作成することができます。距離を測定したい2点の原子の上でそれぞれCtrlキーを押しながらマウスのホイールクリックを行い、コマンドでdistと入力することで距離オブジェクトを生成することができます。