Build
Buildのメニューからは、PyMOLに内蔵されている化合物のフラグメントやアミノ酸残基のデータを利用して任意の化合物またはポリペプチド鎖の構造モデルを生成することができるようになっています。主な利用方法としては、既存のタンパク質構造をロードしたあとに、その末端に長い人工ペプチドを取り付けたモデル構造を作成することや、0から任意のポリペプチド鎖を作成して計算科学の研究の初期構造に利用するという使い方があります。
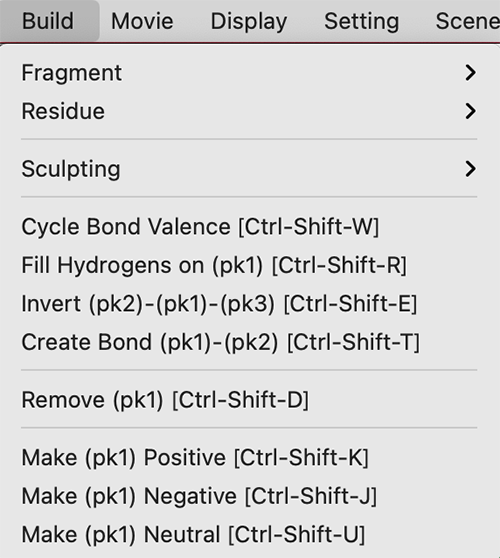
Buildメニューは以下の通りです。
Fragment
ここでは主要な化学小分子の骨格を、フラグメントをつなぎ合わせるような感覚で構築することができます。使い方の例を動画で見てみましょう。
このように、何もない状態から様々な化学骨格を持った分子を作っていくことができます。……ただ、結構癖があって、任意に小分子を作るというのには慣れが必要で、構造最適化部分も含めて他のソフトウェアを使った方がいいような気もします。
やり方としては、まずこのFragmentメニューからベースとなる化合物骨格を選んできます(しかしこの時点でベンゼン環がないので結構アレなんですけれど……。) その後、マウスのPkAt機能(参考:マウス操作の詳細,原子ピッキング)を使って原子を1つピッキングした状態にします。この状態でさらにFragmentメニューからフラグメントを選ぶと、そのピックされた原子部分に選択された化学骨格が生えていきます。これを繰り返していくことで、任意の化合物を作ることが理論上可能です。
化合物の二面角や角度を手動で調整したい場合は、マウス操作で調節して作り出してください(参考:マウス操作の詳細,二面角をピッキング)。また、一応お気持ち程度ですが、後で紹介するSculptingという機能を使うと、簡単な化合物の構造最適化をかけてくれて、無理のない構造に落ち着かせることができます。
Residue
このメニューは20種類の正準アミノ酸の残基のデータの他に、N末端キャッピング用のAcetylとC末端キャッピング用のN-Methylが存在しています。キャッピングとはMDシミュレーション上で使われる人工的な修飾で、末端部分の電荷をニュートラルにするために使われます。それ以外の方はあまり利用することがないでしょう。
AltキーとA~Zの文字を組み合わせて入力することでマウスを使わずに簡単にポリペプチド鎖を作成していくことができます。例えば、PyMOLを開き、BuildメニューからResidue -> Helixを選び、Internal GUI画面上でAltキーを押しながらACDVAARHKと連続して押すと
このようにαヘリックス様のタンパク質フラグメントが簡単に生成されました。同様にして、antiparallel, parallelのβシート構造も作り出すことができます。ただしループ構造は自動で生成してくれませんので、手動で任意の二面角を調節して作り出してください。(参考:マウス操作の詳細,二面角をピッキング)
また、プログラミングしたいときに便利な点として、cmd._alt(chr(XX))を入力することでそのキーを入力した扱いにすることができます。XXにはASCII CODEが入ります(A: 65, B: 66, C: 67, ... 88: X, Y: 89, Z: 90)。
| 表示 | 残基名 | cmd | ショートカットキー |
|---|---|---|---|
| Acetyl | アセチル基 | cmd._alt(chr(66)) | Alt-B |
| Alanine | アラニン | cmd._alt(chr(65)) | Alt-A |
| Amine | アミン | - | |
| Aspartate | アスパラギン酸 | cmd._alt(chr(68)) | Alt-D |
| Asparagine | アスパラギン | cmd._alt(chr(78)) | Alt-N |
| Arginine | アルギニン | cmd._alt(chr(82)) | Alt-R |
| Cysteine | システイン | cmd._alt(chr(67)) | Alt-C |
| Glutamate | グルタミン酸 | cmd._alt(chr(69)) | Alt-E |
| Glutamine | グルタミン | cmd._alt(chr(81)) | Alt-Q |
| Glycine | グリシン | cmd._alt(chr(71)) | Alt-G |
| Histidine | ヒスチジン | cmd._alt(chr(72)) | Alt-H |
| Isoleucine | イソロイシン | cmd._alt(chr(73)) | Alt-I |
| Leucine | ロイシン | cmd._alt(chr(76)) | Alt-L |
| Lysine | リジン | cmd._alt(chr(75)) | Alt-K |
| Methionine | メチオニン | cmd._alt(chr(77)) | Alt-M |
| N-Methyl | N-メチル | cmd._alt(chr(90)) | Alt-Z |
| Phenylalanine | フェニルアラニン | cmd._alt(chr(70)) | Alt-F |
| Proline | プロリン | cmd._alt(chr(80)) | Alt-P |
| Serine | セリン | cmd._alt(chr(83)) | Alt-S |
| Threonine | スレオニン | cmd._alt(chr(84)) | Alt-T |
| Tryptophan | トリプトファン | cmd._alt(chr(87)) | Alt-W |
| Tyrosine | チロシン | cmd._alt(chr(89)) | Alt-Y |
| Valine | バリン | cmd._alt(chr(86)) | Alt-V |
Sculpting
Sculptingは「彫刻する」というような意味ですが、PyMOLのこの機能では、上で生成したような小分子またはポリペプチド鎖について簡単な構造最適化をかけてくれるようです。
ただし、この機能は現在PyMOLの開発者としてはサポートの対象外とされているようで、現時点ではこの機能が改善されることや発見されたバグの修正が行われることはないとされています(参考:PyMOL: Unsupported Features)。したがって、ここでは簡単な使い方の紹介を留める程度にしておきます。
まずSculptingメニューの先にあるサブメニューの2段めにあるSculptingにチェックを入れます(この操作はコマンドでset sculpting, onとすることでもできます)。そしてその下にあるactivateをクリックすると、表示されている分子の構造最適化が自動的に始まります。ただし、この操作は画面に表示されている分子に対して行われてしまうことに注意してください。コマンドではsculpt_activate all とします。途中で止めたい場合はsculpt_deactivate allと入力します。allの部分を生体分子のオブジェクト名にすればその分子のみを構造最適化させることができます。
Cycle Bond Valence [Ctrl-Shift-W]
現在選択中の「結合」に対して、その結合を芳香環のような結合として設定させます。ショートカットキーはCtrl(Macの場合はCommandでも可)とShift-Wの同時押しです。
結合を選択する方法はマウス操作の詳細,二面角をピッキングと同じです。この機能で結合を選択したあと、Ctrl-Shift-Wキーを同時押しすることで、その結合が芳香環様結合として設定させることができます。例えばFragmentメニューに存在するCyclohexyl[Alt-6]にこの機能を適用してベンゼン環に変更したいという場合、6つすべての炭素-炭素結合についてこの操作を行う必要があります。このとき水素原子は自動で増減します。こののち、上記のSculptingによる構造最適化を行えばすべての炭素原子が同一平面上に位置するようになったベンゼン環構造が得られます(が、水素原子の位置がきれいな構造じゃない気がします)。
Fill Hydrogens on (Pk1) [Ctrl-Shift-R]
Pk1で選択されている「原子」に対して、その原子につながっている結合数に応じて水素原子を取り付けてくれます。ショートカットキーはCtrl(Macの場合はCommandでも可)とShift-Rの同時押しです。コマンドはh_fillです。
invert (pk2)-(pk1)-(pk3) [Ctrl-Shift-E]
PkAt機能で原子を3つ選んだ状態、すなわちPk1, Pk2, Pk3が存在している状態でこのコマンドを選択すると、Pk1から先のフラグメントがPk1-Pk2軸回りに180度反転した位置に変化します。ショートカットキーはCtrl(Macの場合はCommandでも可)とShift-Eの同時押しです。コマンドはinvertです。
create bond (pk1)-(pk2) [Ctrl-Shift-T]
PkAt機能で原子を2つ選んだ状態、すなわちPk1, Pk2が存在する状態でこのコマンドを選択すると、2原子の間に結合が形成されます。ショートカットキーはCtrl(Macの場合はCommandでも可)とShift-Tの同時押しです。内部コマンドの処理としてはbond ; unpickが働いています。
これは環を閉じたいときに有用だったり、描画上の問題で離れている2原子を結合したような状態に見せたいときに有用です。
ちなみにコマンド専用ですが、bondコマンドを使うときにbond order=2とすると二重結合として結合を表示させられます。三重結合のときはbond order=3、芳香環様結合のときはbond order=4とします。
Remove (pk1) [Ctrl-Shift-D]
Pk1で選択されている原子を削除します。また、PkTbで選択されている結合についても削除することができます。ショートカットキーはCtrl(Macの場合はCommandでも可)とShift-Dの同時押しです。コマンドはremove_pickedです。
Make (Pk1) positive [Ctrl-Shift-K] / Make (Pk1) negative [Ctrl-Shift-J] / Make (Pk1) neurtral [Ctrl-Shift-U]
Pk1で選択されている原子の電荷を+1, -1, 0にそれぞれ設定させることができます。設定値はLabel機能のother properties, formal chargeを押すと確認することができます。